Tần số xuất hiện vibrio cholerae trên tôm và nhuyễn thể, xác định serogroup O1, O139 và biotype của V. Cholerae bằng kỹ thuật Multiplex-PCR
TÓM TẮT
Nghiên cứu được thực hiện trên 240 mẫu tôm và nhuyễn thể thu thập ở Đồng Nai,
TP. Hồ Chí Minh. Mẫu được tăng sinh bằng hai dung dịch muối pepton kiềm không bổ
sung và bổ sung polymycin B 50 UI và 3 % NaCl. DNA ly trích trực tiếp từ dung dịch
tăng sinh được kiểm tra bằng 3 phản ứng m–PCR để xác định (i) loài V. cholerae (mPCR1); (ii) serogroup O1, O139 (m-PCR2) và (iii) biotype (m-PCR3). Bên cạnh đó,
các gốc vi khuẩn cũng định danh bằng phương pháp sinh hóa và xác định lại bằng các
phản ứng m-PCR.
Phản ứng m-PCR1 phát hiện V. cholerae trên 44,2% số mẫu ở môi trường 1 và
45,8% số mẫu ở môi trường 2. Trong khi đó, phương pháp sinh hóa chỉ phát hiện V.
cholerae ở hai môi trường lần lượt là 41,3% và 10,0%.
Phản ứng m-PCR2 sử dụng DNA ly trích trực tiếp từ dung dịch tăng sinh, kết quả
phát hiện gen từ 106 mẫu ở môi trường 1 và 110 mẫu ở môi trường 2 lần lượt là 2 và 2
mẫu dương tính đồng thời hai gen ctxA và rfbO1, 8 và 11 mẫu dương tính đồng thời hai
gen ctxA và rfbO139, 2 và 4 mẫu chỉ dương tính với rfbO1, 2 và 4 mẫu chỉ dương tính
với rfbO139, 13 và 18 mẫu chỉ dương tính với ctxA. Ngoài ra, môi trường tăng sinh 2
còn giúp phát hiện được hai mẫu dương tính đồng thời cả 3 gen ctxA, rfbO1 và
rfbO139.
Sử dụng DNA của từng gốc đã định danh, kết quả m-PCR2 phát hiện serogroup
từ 99 mẫu ở môi trường 1 và 24 mẫu ở môi trường 2 lần lượt là 5 và 3 mẫu dương tính
với V. cholerae O139 sinh độc tố CT, 11 và 5 mẫu dương tính với V. cholerae
O1/O139 không sinh độc tố CT, 5 và 2 mẫu dương tính với V. cholerae non – O1/O139
sinh độc tố CT.
Kết quả phản ứng m-PCR3 cho thấy chỉ 3 trong 6 mẫu phát hiện được gen tcpA
đặc trưng cho V. cholerae O1 biotype El Tor.
Một số gốc V. cholerae phân lập được đều nhạy cảm doxycycline và norfloxacin
và đề kháng hoàn toàn với colistine, đề kháng cao với oxacillin

Trang 1

Trang 2

Trang 3
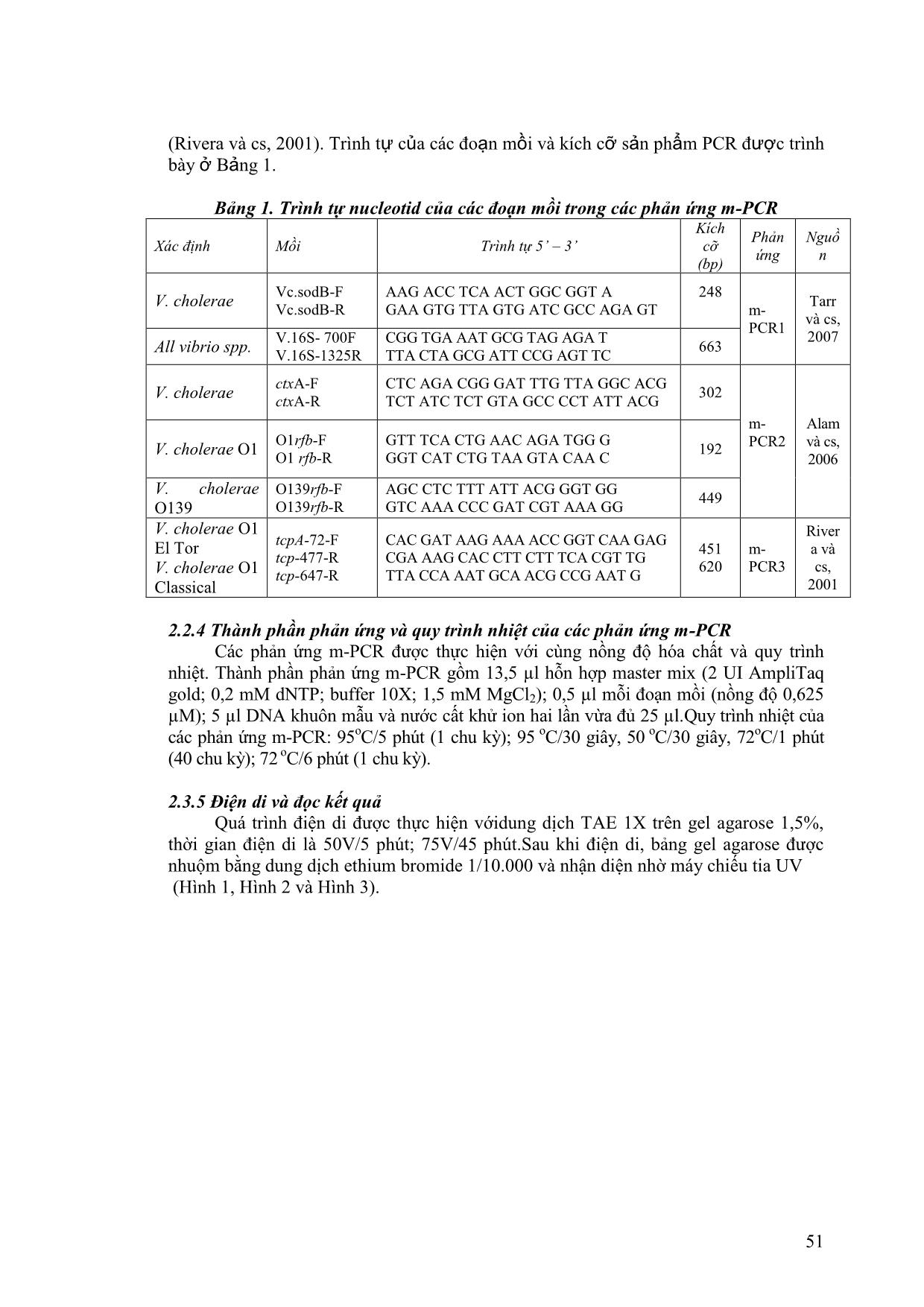
Trang 4
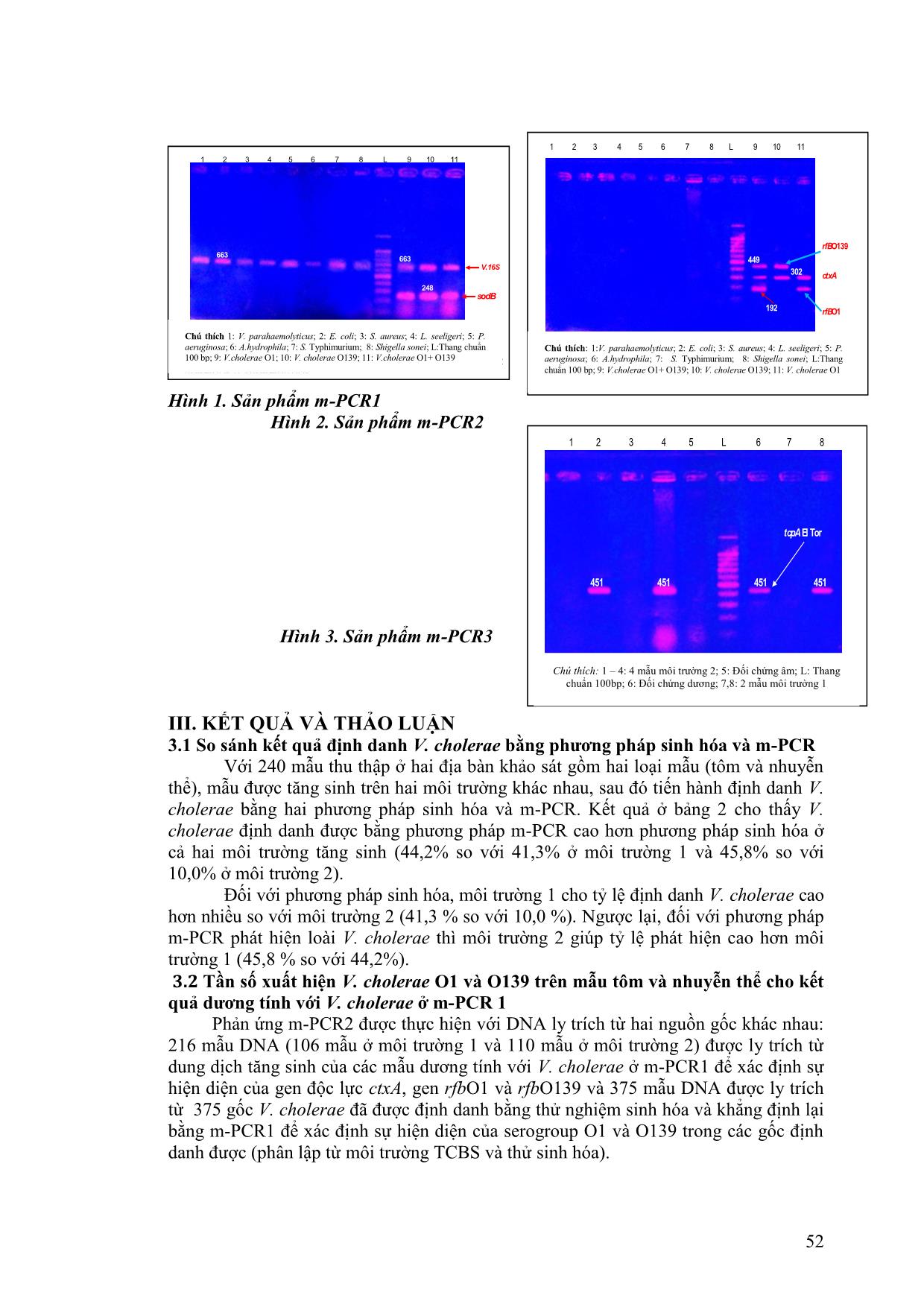
Trang 5
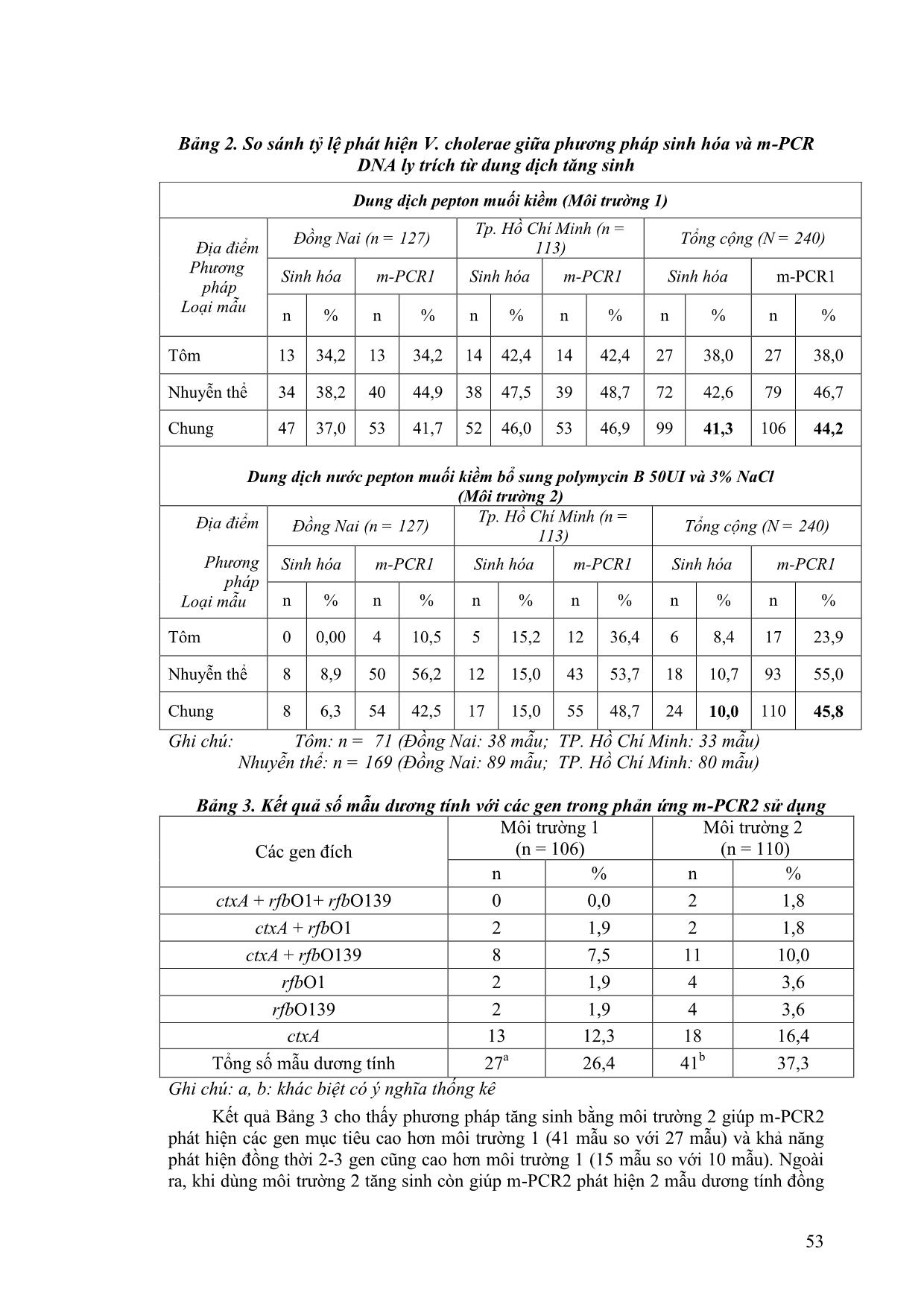
Trang 6

Trang 7

Trang 8

Trang 9

Trang 10
Tóm tắt nội dung tài liệu: Tần số xuất hiện vibrio cholerae trên tôm và nhuyễn thể, xác định serogroup O1, O139 và biotype của V. Cholerae bằng kỹ thuật Multiplex-PCR

ng tính với V. cholerae ở m-PCR 1 Phản ứng m-PCR2 được thực hiện với DNA ly trích từ hai nguồn gốc khác nhau: 216 mẫu DNA (106 mẫu ở môi trường 1 và 110 mẫu ở môi trường 2) được ly trích từ dung dịch tăng sinh của các mẫu dương tính với V. cholerae ở m-PCR1 để xác định sự hiện diện của gen độc lực ctxA, gen rfbO1 và rfbO139 và 375 mẫu DNA được ly trích từ 375 gốc V. cholerae đã được định danh bằng thử nghiệm sinh hóa và khẳng định lại bằng m-PCR1 để xác định sự hiện diện của serogroup O1 và O139 trong các gốc định danh được (phân lập từ môi trường TCBS và thử sinh hóa). sodB V.16S 1 2 3 4 5 6 7 8 L 9 10 11 Chú thích 1: V. parahaemolyticus; 2:E. coli; 3: S. aureus; 4: L. seeligeri; 5: P. aeruginosa; 6: A.hydrophila; 7: S. typhimurium; 8: Shigella sonei; L:Thang chuẩn 100 bp; 9: V.cholerae O1; 10: V. cholerae O139; 11: V.cholerae O1+ O139. 248 663 663 Chú thích:1 – 5: 5 mẫu môi trường 2; L: Thang chuẩn 100 bp; 6: Đối chứng dương; 7 và 8: mẫu khảo sát môi trường 1 1 2 3 4 5 L 6 7 8 451 451 451 451 tcpA El Tor Chú thích: 1 – 4: 4 mẫu môi trường 2; 5: Đối chứng âm; L: Thang chuẩn 100bp; 6: Đối chứng dương; 7,8: 2 mẫu môi trường 1 Chú thích:1:V. parahaemolyticus; 2:E. coli; 3: S. aureus; 4: L. seeligeri; 5: P. aeruginosa; 6: A.hydrophila; 7: S. typhimurium; 8: Shigella sonei; L:Thang chuẩn 100 bp; 9: V.cholerae O1+ O139; 10: V. cholerae O139;11: V. cholerae O1 302 rfBO1 449 192 ctxA rfBO139 1 2 3 4 5 6 7 8 L 9 10 11 Chú thích: 1:V. parahaemolyticus; 2: E. coli; 3: S. aureus; 4: L. seeligeri; 5: P. aeruginosa; 6: A.hydrophila; 7: S. Typhimurium; 8: Shigella sonei; L:Thang chuẩn 100 bp; 9: V.cholerae O1+ O139; 10: V. cholerae O139; 11: V. cholerae O1 Chú thích 1: V. parahaemolyticus; 2: E. coli; 3: S. aureus; 4: L. seeligeri; 5: P. aeruginosa; 6: A.hydrophila; 7: S. Typhimurium; 8: Shigella sonei; L:Thang chuẩn 100 bp; 9: V.cholerae O1; 10: V. cholerae O139; 11: V.cholerae O1+ O139 53 Bảng 2. So sánh tỷ lệ phát hiện V. cholerae giữa phương pháp sinh hóa và m-PCR DNA ly trích từ dung dịch tăng sinh Các gen đích Môi trường 1 (n = 106) Môi trường 2 (n = 110) n % n % ctxA + rfbO1+ rfbO139 0 0,0 2 1,8 ctxA + rfbO1 2 1,9 2 1,8 ctxA + rfbO139 8 7,5 11 10,0 rfbO1 2 1,9 4 3,6 rfbO139 2 1,9 4 3,6 ctxA 13 12,3 18 16,4 Tổng số mẫu dương tính 27a 26,4 41b 37,3 Ghi chú: a, b: khác biệt có ý nghĩa thống kê Kết quả Bảng 3 cho thấy phương pháp tăng sinh bằng môi trường 2 giúp m-PCR2 phát hiện các gen mục tiêu cao hơn môi trường 1 (41 mẫu so với 27 mẫu) và khả năng phát hiện đồng thời 2-3 gen cũng cao hơn môi trường 1 (15 mẫu so với 10 mẫu). Ngoài ra, khi dùng môi trường 2 tăng sinh còn giúp m-PCR2 phát hiện 2 mẫu dương tính đồng Dung dịch pepton muối kiềm (Môi trường 1) Địa điểm Phương pháp Loại mẫu Đồng Nai (n = 127) Tp. Hồ Chí Minh (n = 113) Tổng cộng (N = 240) Sinh hóa m-PCR1 Sinh hóa m-PCR1 Sinh hóa m-PCR1 n % n % n % n % n % n % Tôm 13 34,2 13 34,2 14 42,4 14 42,4 27 38,0 27 38,0 Nhuyễn thể 34 38,2 40 44,9 38 47,5 39 48,7 72 42,6 79 46,7 Chung 47 37,0 53 41,7 52 46,0 53 46,9 99 41,3 106 44,2 Dung dịch nước pepton muối kiềm bổ sung polymycin B 50UI và 3% NaCl (Môi trường 2) Địa điểm Phương pháp Loại mẫu Đồng Nai (n = 127) Tp. Hồ Chí Minh (n = 113) Tổng cộng (N = 240) Sinh hóa m-PCR1 Sinh hóa m-PCR1 Sinh hóa m-PCR1 n % n % n % n % n % n % Tôm 0 0,00 4 10,5 5 15,2 12 36,4 6 8,4 17 23,9 Nhuyễn thể 8 8,9 50 56,2 12 15,0 43 53,7 18 10,7 93 55,0 Chung 8 6,3 54 42,5 17 15,0 55 48,7 24 10,0 110 45,8 Ghi chú: Tôm: n = 71 (Đồng Nai: 38 mẫu; TP. Hồ Chí Minh: 33 mẫu) Nhuyễn thể: n = 169 (Đồng Nai: 89 mẫu; TP. Hồ Chí Minh: 80 mẫu) Bảng 3. Kết quả số mẫu dương tính với các gen trong phản ứng m-PCR2 sử dụng 54 thời gen ctxA cùng với rfbO1 và rfbO139 mà ở môi trường 1 không giúp phát hiện được; hoặc 2 mẫu ở môi trường 1 so với 4 mẫu ở môi trường 2 đối với gen rfbO1; hoặc 2 mẫu ở môi trường 1 so với 4 mẫu ở môi trường 2 đối với gen rfbO139, hoặc 13 mẫu ở môi trường 1 so với 18 mẫu ở môi trường 2 đối với gen ctxA. Kết quả dương tính đồng thời của gen ctxA và gen rfbO1 hoặc/ và rfbO139 cho thấy khả năng hiện diện các V. cholerae mang gen độc lực trong mẫu khảo sát. Nhằm xác định sự hiện diện của các serogroup này trong mẫu khảo sát, m-PCR2 được tiến hành với DNA của 375 gốc (tương ứng 123 mẫu thực địa) V. cholerae đã được định danh (Bảng 4). Bảng 4. Tần số xuất hiện V. cholerae O1 và O139 trên mẫu khảo sát (DNA của từng gốc định danh bằng sinh hóa) Serogroup của V. cholerae Môi trường 1 (n = 99) Môi trường 2 (n = 24) n % n % V. cholerae O1 có độc tố CT (ctxA + rfbO1) 0 0,0 0 0,0 V. cholerae O139 có độc tố CT (ctxA + rfbO139) 5 5,0 3 9,4 V. cholerae O1 không có độc tố CT (rfbO1) 3 3,0 2 8,3 V. cholerae O139 không có độc tố CT (rfbO139) 8 8,1 3 12,5 V. cholerae non – O1/139 có độc tố (ctxA) 5 5,0 2 8,3 Kết quả Bảng 4 cho thấy 5 mẫu ở môi trường 1 và 3 mẫu ở môi trường 2 được xác định sự hiện diện của V. cholerae serogroup O139 có độc tố CT, không có mẫu nào phát hiện được V. cholerae serogroup O1 sinh độc tố CT. Ngoài ra, có 3 mẫu ở môi trường 1 và 2 mẫu môi trường 2 hiện diện V. cholerae serogroup O1 không sinh độc tố CT; 8 mẫu ở môi trường 1 và 3 mẫu môi trường 2 hiện diện V. cholerae serogroup O139 không sinh độc tố CT; 5 mẫu ở môi trường 1 và 2 mẫu môi trường 2 hiện diện V. cholerae thuộc các serogroup non – O1/ non – O139 sinh độc tố CT. 3.3 Xác định biotype của V. cholerae O1 trên mẫu tôm và nhuyễn thể cho kết quả dƣơng tính với V. cholerae O1 ở m-PCR 2 DNA của các mẫu dương tính đồng thời ít nhất hai gen ctxA và rfbO1 ở phản ứng m-PCR2 (2 mẫu ở môi trường 1 và 4 mẫu ở môi trường 2, xem Bảng 3) được sử dụng để xác định biotype của V. cholerae O1. Kết quả cho thấy 1 mẫu ở môi trường 1 và 2 mẫu ở môi trường 2 cho kết quả dương tính với gen tcpA đặc trưng cho V. cholerae O1 biotype El Tor, không mẫu nào cho kết quả với tcpA đặc trưng cho V. cholerae O1 biotype cổ điển (Hình 3). Thảo luận Phương pháp PCR cho kết quả phát hiện V. cholerae chính xác và nhạy hơn nhiều so với phương pháp sinh hóa. Lý do là phương pháp PCR có thể phát hiện được các dòng V. cholerae tồn tại hoặc không tồn tại trên môi trường phân lập (Lipp và cs, 2003). Bên cạnh đó, phương pháp PCR sử dụng DNA tách chiết từ dung dịch tăng sinh sẽ cho kết quả phát hiện V. cholerae nhanh hơn, chính xác và độ nhạy cao hơn so với phương pháp nuôi cấy truyền thống (Lipp và cs, 2003). Tuy vậy, khi sử dụng DNA tách chiết từ dung dịch tăng sinh để thực hiện phản ứng m-PCR thì không thể khẳng định sự tồn tại của các serogroup của V. cholerae mang gen độc lực mà chỉ cho thấy nguy cơ hiện diện của các dòng vi khuẩn này trong mẫu khảo sát. Tần số xuất hiện của V. cholerae trong mẫu tôm và nhuyễn thể ở hai địa bàn khảo sát khá cao (Bảng 2). Tuy nhiên, khả năng xuất hiện của các serogroup mang gen độc lực của V. cholerae trong mẫu khảo sát rất thấp (Bảng 3 và Bảng 4). Điều đó cho thấy các serogroup của V. cholerae tồn tại trong mẫu khảo sát cũng như trong môi 55 trường chủ yếu là non-O1, O139 (Goel và cs, 2010) hoặc những dòng V. cholerae O1 và O139 không sinh độc tố CT (Kaper và cs, 1995). Tỷ lệ phát hiện gen sodB của V. cholerae trong phân vật nuôi là 32% (74/230 mẫu), trong số này, chỉ phát hiện 20/74 mẫu có mang gen rfb của V. cholerae O1 và không có mẫu nào dương tính với V. cholerae O139 (Keshav và cs, 2010). Chúng tôi tiến hành định biotype của 6 mẫu cho kết quả dương tính đồng thời với ít nhất 2 gen của V. cholerae O1, nhưng chỉ có 3 mẫu cho kết quả dương tính với gen tcpA (Hình 3). Thật vậy, trái với nghiên cứu trước đây cho rằng hầu hết các dòng V. cholerae O1 đều cho kết quả dương tính với ctxA và tcpA (Faruque và cs, 1998), 37% số mẫu được xác định là V. cholerae O1 đã không hiện diện gen ctxA và tcpA (Aulet và cs, 2007). Ngoài ra, một số nghiên cứu đã chứng minh một số dòng Vibrio cholerae non-O1/ non-O139 cũng có khả năng mang đồng thời hai gen độc lực ctxA và tcpA (Singh và cs, 2001). Kết quả nghiên cứu cho thấy không có mẫu nào (dung dịch tăng sinh hoặc gốc phân lập được) cho kết quả dương tính với V. cholerae O1 biotype cổ điển. Từ năm 1961, V. cholerae O1 biotype El Tor đã thay thế V. cholerae O1 biotype cổ điển trong các ổ dịch tả (Alm và Manning, 1990). Hầu hết các gốc phân lập từ Việt Nam và Bangladesh đều là El Tor (Minh và cs, 2009). Tuy nhiên, không phải tất cả các nghiên cứu đều cho thấy V. cholerae O1 biotype cổ điển đã biến mất mà chúng vẫn đang tồn tại. Nghiên cứu của Safa và cs (2008) khảo sát 41 dòng V. cholerae O1 phân lập ở châu Á và châu Phi từ năm 1991 đến năm 2004 cho thấy tất cả các dòng đều cho kết quả dương tính với gen rfb O1, hầu hết mang gen tcpA đặc hiệu cho biotype cổ điển (451bp) ngoại trừ 5 dòng ở Việt Nam đều mang tcpA El Tor. 3.3 Kết quả kháng sinh đồ của V. cholerae định danh đƣợc Nghiên cứu đã tiến hành thử kháng sinh đồ để đánh giá tính nhạy cảm của 9 gốc V. cholerae định danh được đối với các loại kháng sinh thường sử dụng để điều trị bệnh tả. Kết quả kháng sinh đồ của các gốc V. cholerae định danh được thể hiện qua Bảng 5. Bảng 5. Tính nhạy cảm của V. cholerae với một số loại kháng sinh (n = 9) Kháng sinh Nhạy Trung gian Kháng n % n % n % Amoxicillin 3 33,3 2 22,2 4 44,4 Cloramphenicol 7 77,8 1 11,1 1 11,1 Colistine 0 0,0 0 0,0 9 100,0 Doxycycline 9 100,0 0 0,0 0 0,0 Gentamicin 8 88,9 0 0,0 1 11,1 Norfloxacin 9 100,0 0 0,0 0 0,0 Oxacillin 1 11,1 0 0,0 8 88,9 Polymycin B 3 33,3 0 0,0 6 66,7 Trimethoprim/sulfamethoxazol 4 44,4 0 0,0 5 55,6 Vancomycin 2 22,2 3 33,3 4 44,4 Kết quả cho thấy V. cholerae nhạy cảm hoàn toàn với doxycycline và norfloxacin (100%); khá nhạy cảm với gentamicin (88,9%) và cloramphenicol (77,8%); đề kháng hoàn toàn với colistine (100%) và đề kháng cao với oxacillin (88,9%) và tương đối kháng polymycin B (66,7%), vancomycin (44,4%) và trimethoprim/sulfamethoxazole (55,6%). Vấn đề đề kháng kháng sinh của V. cholerae đã được rất nhiều tác giả nghiên cứu trước đây, kết quả cho thấy khả năng đề kháng của vi khuẩn này rất đa dạng, tùy thuộc vào thời gian và địa điểm nghiên cứu. Ở nước ta, các dòng V. cholerae O1/non- O1 từ năm 1995- 2002 đề kháng với streptomycin, sulfamethoxazole/trimethoprim 56 (Ehara và cs, 2004), các dòng phân lập năm 2003 thì đề kháng với amoxicillin và erythromycin (Bani và cs, 2007). V. cholerae phân lập ở Bangladesh 2002-2008 đề kháng với tetracycline, ciprofloxacin, sulfamethoxazole/trimethoprim, erythromycin và furazolidone (Kim và cs, 2010). Theo Ang và cs (2010) các dòng V. cholerae O1 El Tor phân lập được ở Malaysia năm 2009 có khả năng đề kháng với nhiều loại kháng sinh bao gồm tetracycline, erythromycin, streptomycin, penicillin G, sulfamethoxazole/trimethoprim và polymycin B. Gần đây nhất, V. cholerae O1 ở Indonesia đề kháng với erythromycin nhưng nhạy cảm với nhiều loại kháng sinh ampicillin, ciprofloxacin, cloramphenicol, tetracycline, gentamicin, kanamicin, sulfamethoxazole/trimethoprim, norfloxacin, streptomycin và axit nalidixic (Nishibori và cs, 2011). IV KẾT LUẬN Môi trường nước muối pepton kiềm là môi trường tăng sinh hiệu quả để định danh V. cholerae bằng phương pháp sinh hóa, bổ sung polymycin B và 3% NaCl vào môi trường này rất có ích cho việc phát hiện các gen độc lực của loài vi khuẩn này bằng phương pháp m-PCR. Tần số xuất hiện của V. cholerae trong mẫu tôm và nhuyễn thể khá cao (hơn 40%) nhưng sự hiện diện của các serogroup O1, O139 sản sinh độc tố CT trong mẫu khảo sát rất thấp (5 mẫu ở môi trường 1 và 3 mẫu ở môi trường 2). Chỉ phát hiện được gen tcpA đặc trưng cho V. cholerae O1 biotype El Tor ở 3/6 mẫu khảo sát, không mẫu nào cho kết quả dương tính với tcpA đặc trưng cho V. cholerae O1 biotype cổ điển. Một số gốc V. cholerae phân lập được đều nhạy cảm doxycycline và norfloxacin và đề kháng hoàn toàn với colistine, đề kháng cao với oxacillin. TÀI LIỆU THAM KHẢO 1.Alam M., Sultana M., Nair G. B., Sack R. B., Sack D. A., Siddique A. K., Ali A., Huq A. and Colwell R. R., 2006. Toxigenic Vibrio cholerae in the aquatic environment of Mathbaria, Bangladesh. Applied and Environmental Microbiology 72 (4): 2849- 2855. 2.Ang G. Y., Yu C. Y., Balqis K., Elina H. T., Azura H., Hani M. H. and Yean C. Y., 2010. Molecular evidence of cholera outbreak caused by a toxigenic Vibrio cholerae O1 El Tor variant strain in Kelantan, Malaysia. Journal of Clinical Microbiology 48 (11): 3963-3969. 3.Bani S., Mastromarino P. N., Ceccarelli D., An L. V., Salvia A. M., Tram N. V. Q., Hai D. H., Bacciu D., Cappuccinelli P. and ColomboM. M., 2007. Molecular characterization of Vibrio cholerae and its disappearance in Vibrio cholerae O1 strains isolated in 2003 in Vietnam. FEMS Microbiology Letters 266: 42-48. 4.Ehara M., Nguyen B. M., Nguyen D. T., Toma C., Higa N. And Iwanaga M., 2004. Drug susceptibility and its genetic basis in epidemic Vibrio cholerae O1 in Vietnam. Epidemiology and Infection 132: 595-600. 5.Faruque S. M., Albert M. J. and Mekalanos J. J., 1998. Epidemiology, genetics and ecology of toxigenic Vibrio cholerae.Microbiology and Molecular Biology Reviews 62(4): 1301-1314. 6.Kim H. B., Wang M.,Ahmed S., ParkC.H., LaRocque R. C.,Faruque A. S., Salam M. A., Khan W. A., Qadri F., Calderwood S. B., Jacoby G. A. and Hooper D. C., 2010.Transferable quinolone resistance in Vibrio cholerae. Antimicrobial Agents and Chemotherapy 54 (2): 799-803. 57 7.Minh N. B., Lee H. J., Cuong N. T., Choi Y. S., Hien N. T., Anh D. D., Lee R. H., Ansaruzzaman M., Endtz P. H., Chun J., Lopez L. A., Czerkinsky C., Clemens D. J. and Kim W. D., 2009. Cholera outbreaks caused by an altered Vibrio cholerae O1 EI Tor biotype strain producing classical cholera toxin B in Vietnam in 2007 to 2008. Journal of Clinical Microbiology 47(5): 1568-1571. 8.Nishibori T.,Cores de Vries G., Rahardjo D., Wasito E. B., De I., Kinoshita S., Hayashi Y., Hotta H., Kawabata M., Shirakawa T., Iijima Y. and Osawa R., 2011. Phenotypic and genotypic characterization of Vibrio cholerae clinically isolated in Surabaya, Indonesia. Indonesia – Japan journal infectious diseases 64: 7-12. 9.Singh D. V., Matte M., Matte g. R., Jiang S., Sabeena F., Shukla B. N., Sanyal S. C., Huq A. and Colwell R. R., 2001. Molecular analysis of Vibrio cholerae O1, O139, non – O1, and non – O139 strains: clonal relationships between clinical and environmental isolates. Applied and Environmental Microbiology 67 (2): 910-921 10.Tarr C. L., Patel S. J., Puhr N. D., Sowers E. G., Bopp C. A. and Strockbine N. A., 2007. Identification of Vibrio isolates by a multiplex PCR assay and rpoB sequence determination. Journal of Clinical Microbiology 45 (1): 134-140.
File đính kèm:
 tan_so_xuat_hien_vibrio_cholerae_tren_tom_va_nhuyen_the_xac.pdf
tan_so_xuat_hien_vibrio_cholerae_tren_tom_va_nhuyen_the_xac.pdf

