Quy trình phân tích đoạn cytochrome B trên các mẫu cá tra (Pangasianodon hypophthalmus)
TÓM TẮT
Với mục đích kiểm định cá Tra (Pangasianodon hypophthalmus), việc xây dựng quy trình phân
tích đoạn Cytochrome b là cần thiết. Cặp mồi universal cho Cytb được sử dụng để nhân gen thuộc
vùng gen ty thể thông qua PCR các mẫu cá Tra và một số loài cá da trơn khác với kích thước 430
bp. Sản phẩm PCR sau đó được tinh sạch và giải trình tự. Các chuỗi trình tự nghiên cứu sau khi giải
cần phải được xử lý sơ bộ trước khi được đưa vào chương trình phân tích chính. Đầu tiên, các chuỗi
trình tự cần được kiểm tra chất lượng giải trình tự và xử lý bằng kết hợp hai phần mềm FinchTV và
Bioedit để đạt được trình tự chuẩn xác nhất cho các bước phân tích tiếp theo. Kế tiếp, dùng phần
mềm BLAST của NCBI để so sánh và xác định mức độ tương đồng của mẫu nghiên cứu với trình
tự chuẩn đã được công bố trong dữ liệu của NCBI. Cuối cùng, phần mềm Mega 6 sẽ là phần mềm
thích hợp cho các phân tích về sự phát sinh loài và khoảng cách di truyền của cá Tra và những loài
cá da trơn khác trong họ Pangasiidae muốn quan tâm.

Trang 1

Trang 2

Trang 3

Trang 4

Trang 5

Trang 6

Trang 7
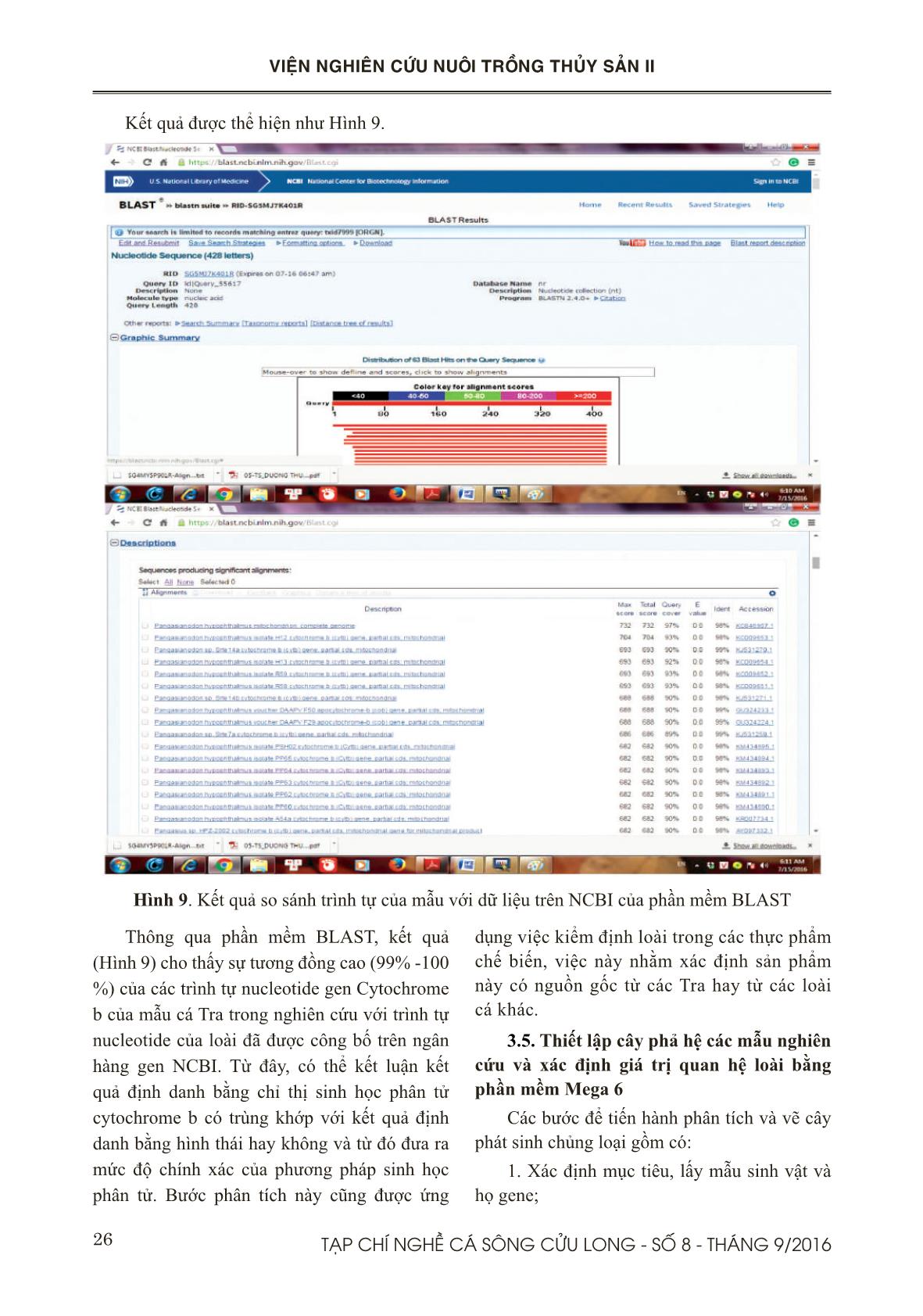
Trang 8
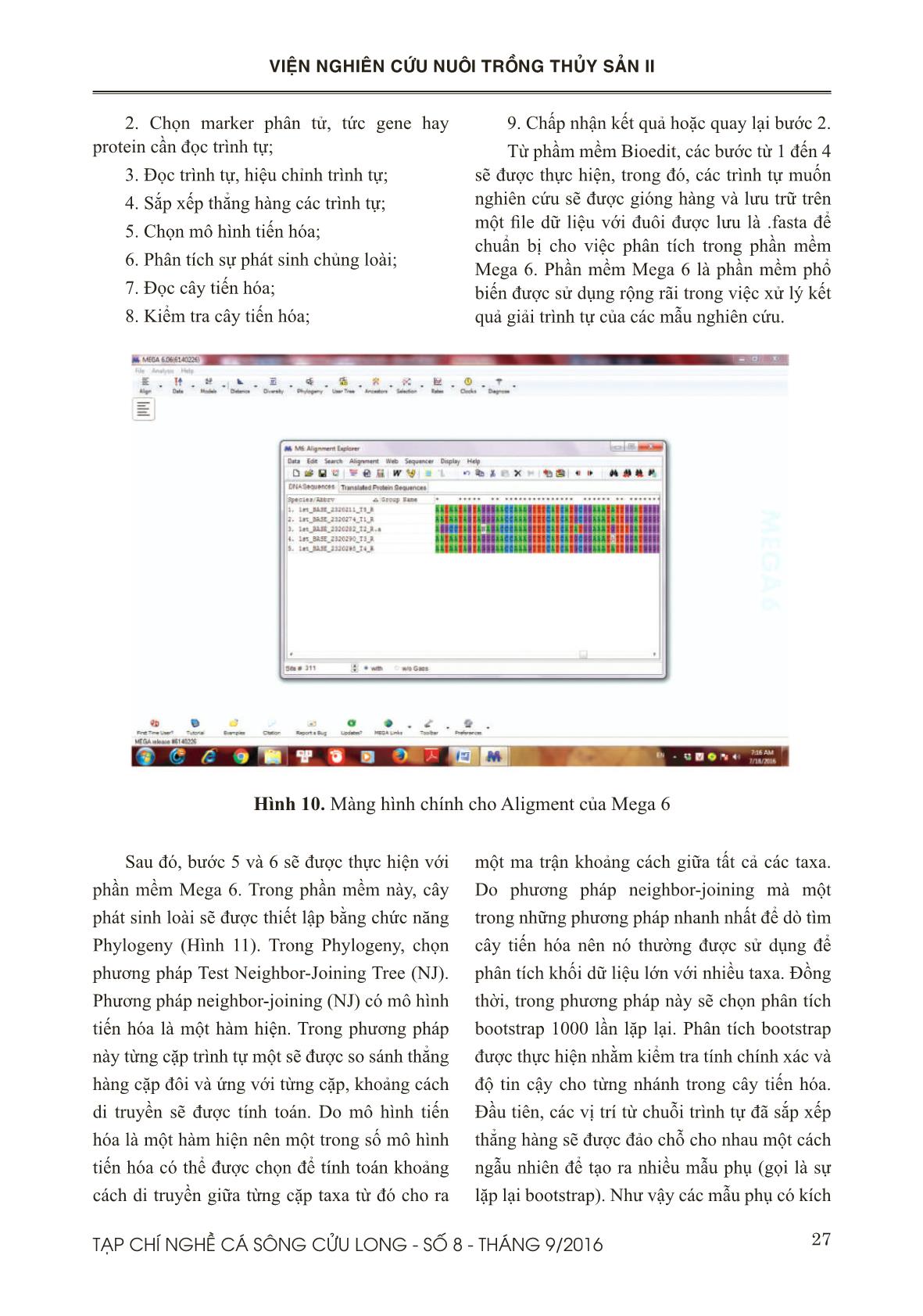
Trang 9

Trang 10
Tải về để xem bản đầy đủ
Tóm tắt nội dung tài liệu: Quy trình phân tích đoạn cytochrome B trên các mẫu cá tra (Pangasianodon hypophthalmus)

ụng BLAST để so sánh trình tự được đưa vào cơ sở dữ liệu của trình tự đáp ứng công ước quốc tế dựa vào tiêu chuẩn cho mã vạch DNA Cytochrome b Bước 2: Chọn “BLAST” từ menu ở phía phải của trang chủ để thực hiện bước tìm kiếm dữ liệu và gióng hàng với trình tự gốc của dữ liệu NCBI Bước 3: Chọn “Nucleotide Blast” từ màn hình chính của BLAST (Hình 2.) Hình 2: Màn hình chính của phần mềm BLAST, NCBI 23TẠP CHÍ NGHỀ CÁ SÔNG CỬU LONG - SỐ 8 - THÁNG 9/2016 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN II Bước 4: Dán chuỗi trình tự vào mục “Enter query sequence in fasta format” và kích “BLAST”. Bước 5: Nhìn vào kết quả tìm kiếm NCBI cho biết những loài gì được tìm thấy gần nhất với đoạn trình tự được tra tìm. Kết quả cho biết về Mức độ bao phủ (Query cover), mức tương đồng (Identification) với loài gần nhất Bước 6: Kích vào nút “Taxonomy reports ” và “Distance tree of results”trên trang web để xem báo cáo về phân loại và cây phát sinh chủng loại mà cơ sở dữ liệu NCBI xây dựng. 2.2.8. Thiết lập cây phả hệ các mẫu nghiên cứu và xác định giá trị quan hệ loài Dữ liệu trình tự Cytochrome b của cả mẫu mới giải mã và dữ liệu có sẵn trong ngân hàng gen sẽ được chuẩn bị để phân tích khoảng các di truyền giữa các mẫu bằng cách sử dụng phần mềm MEGA phiên bản 6.0 (Tamura và ctv., 2007). Sử dụng phương pháp Neighbour- joining (NJ) và mô hình khoảng cách Kimura 2-Parameter (K2P) (Kimura, 1980) có trong phần mềm MEGA để xây dựng cây phát sinh chủng loại của Cytochrome b. Với bộ dữ liệu gốc, bootstrap 1000 lần được thực hiện nhằm đánh giá độ tin cậy của sự phân nhánh trên các cây phát sinh chủng loại NJ. Các chỉ số về sự khác biệt trong cùng loài và cùng giống (conspecific and congeneric divergences) sẽ được tính toán. Các dữ liệu được phân nhóm (putative species clusters) dựạ vào cây phát sinh chủng loại NJ. Các nhóm được cho là cùng loài khi có độ chênh lệch hay khác biệt dưới 2%. III. KẾT QUẢ VÀ THẢO LUẬN 3.1. Kết quả tách chiết DNA Để thử nghiệm qui trình tách chiết DNA, chúng tôi tiến hành tách trên 7 mẫu vây cá Tra thu được ở Long Xuyên, tỉnh An Giang. Hình 3. Kết quả kiểm tra chất lượng DNA bằng gel agarose Dựa vào kết quả trên Hình 3, cho thấy DNA tách chiết được với hàm lượng lớn, không bị đứt gãy, sạch và ít bị nhiễm RNA. Ngoài ra, chúng tôi dùng máy đo Nanodrop để kiểm tra một cách chính xác hàm lượng và mức độ tinh sạch cua sản phẩm DNA (Bảng 2). Bảng 2. Kết quả kiểm tra nồng độ DNA bằng máy đo nồng độ Nanodrop Từ kết quả trên ở Bảng 2 cho thấy hàm lượng DNA thu được tương đối cao và chỉ số OD 260/280 điều nhỏ hơn 1,8. Điều này cho thấy phương pháp tách chiết DNA bằng tủa muối hoàn toàn thích hợp cho nghiên cứu của đề tài. 3.2. Phản ứng PCR 3.2.1. Kết quả tối ưu phản ứng PCR Từ kết quả điện di trên gel agarose (Hình 4) cho thấy sản phẩm PCR nằm trong khoảng 430 bp. Sản phẩm PCR tốt ở tất cả hầu hết nhiệt độ lai. Do đó, nhiệt độ Ta thích hợp cho phản ứng khuếch đại sẽ được lựa chọn theo công bố của tác giả 52,6oC. 24 TẠP CHÍ NGHỀ CÁ SÔNG CỬU LONG - SỐ 8 - THÁNG 9/2016 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN II Hình 4. Kết quả tối ưu mồi cytb 3.2.2. Kết quả phản ứng PCR Hình 5. Kết quả chạy PCR với cặp mồi cytochrome b. Kết quả PCR trên một số mẫu cá Tra ngẫu nhiên (Hình 5) cho thấy quy trình chạy của tác giả cũng hoàn toàn thích hợp cho điều kiện phòng thí nghiệm của đề tài mặc dù sản phẩm DNA khuyếch đại có hàm lượng hơi thấp. Tuy nhiên, điều này sẽ được điều chỉnh khi tĕng hàm lượng DNA đầu vào cho phản ứng PCR. 3.3. Phân tích và sắp xếp trình tự bằng phần mềm FinchTV Trình tự nhận được từ công ty First BASE Laboratories, Malaysia có 2 file với hai đuôi AB1 và SEQ, dùng file đuôi AB1 cho phần mềm FinchTV. Từ màn hình chính (Hình 6), nhập “Open” để nhập đoạn trình tự mong muốn kiểm tra chất lượng giải trình tự với đuôi file .AB1. Hình 6. Màn hình chính của phần mềm FinchTV 25TẠP CHÍ NGHỀ CÁ SÔNG CỬU LONG - SỐ 8 - THÁNG 9/2016 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN II Hình 7. Kết quả kiểm tra chất lượng giải trình tự của sản phẩm Theo quy ước, mỗi một màu chỉ thị cho một loại nucleotide, trong đó Adenine (A) = Xanh lá cây, Thymine (T) = đỏ, Cytosine (C) = Xanh dương, Guanine (G) = Đen (Hình 7). Từ kết quả cho thấy, tín hiệu đọc của mỗi nucleotide rõ ràng và tách biệt chứng tỏ kết quả đọc chính xác, rõ ràng, không bị tạp nhiễm. Ngoài ra, giá trị của chất lượng giải trình tự (Quality values) còn được thể hiện khi nhấp vào biểu tượng có hình “Q” trên menu. Sau đó, khi nhấp chuột vào một nucleotide nào đó, ở góc dưới cùng bên trái sẽ hiện ra giá trị Q và vị trí của nucleotide. Ví dụ, khi nhấp chuột vào nucleotide ở vị trí thứ 428, giái trị Q được hiển thị là Q(10), điều này có nghĩa là có nghĩa là nucleotide thứ 428 sẽ có xác suất đọc sai là 1/10. Khi giá trị Q này nằm ở 30 hoặc cao hơn thì việc đọc nucleotide ở vị trí đó được xem là chính xác. Ngoài ra, khi gióng hàng mồi xuôi và mồi ngược, nếu có sự sai khác giữa hai trình tự thì nucleotide sẽ được chọn dựa vào giá trị Q. Dựa vào giá trị Q của FinchTV, chúng ta sẽ biết được bắt đầu vị trí nào sẽ bị cắt bỏ trong đoạn trình tự bằng phần mềm Bioedit nhầm loại bỏ đi đoạn mồi được gắn vào trong qua trình giải trình tự. Đối với dữ liệu nhiều chuỗi trình tự, các trình tự này sẽ được gióng hàng, cắt bỏ hai đầu cho bằng nhau và save lại ở một file dạng fasta chuẩn bị cho việc phân tích sau này. 3.4. Kết quả so sánh trình tự với dữ liệu trên NCBI Những đoạn trình tự sau khi xử lý hoàn chỉnh, được sử dụng để so sánh với trình tự dữ liệu trên NCBI. Các thao tác được thực hiện như sau: dán chuỗi trình tự muốn so sánh vào ô “Enter query sequence”, Chọn “Pangasiidae” trong ô Organism và nhấn nút BLAST (Hình 8). Hình 8. Màn hình chính của phần mềm BLAST 26 TẠP CHÍ NGHỀ CÁ SÔNG CỬU LONG - SỐ 8 - THÁNG 9/2016 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN II Kết quả được thể hiện như Hình 9. Thông qua phần mềm BLAST, kết quả (Hình 9) cho thấy sự tương đồng cao (99% -100 %) của các trình tự nucleotide gen Cytochrome b của mẫu cá Tra trong nghiên cứu với trình tự nucleotide của loài đã được công bố trên ngân hàng gen NCBI. Từ đây, có thể kết luận kết quả định danh bằng chỉ thị sinh học phân tử cytochrome b có trùng khớp với kết quả định danh bằng hình thái hay không và từ đó đưa ra mức độ chính xác của phương pháp sinh học phân tử. Bước phân tích này cũng được ứng dụng việc kiểm định loài trong các thực phẩm chế biến, việc này nhằm xác định sản phẩm này có nguồn gốc từ các Tra hay từ các loài cá khác. 3.5. Thiết lập cây phả hệ các mẫu nghiên cứu và xác định giá trị quan hệ loài bằng phần mềm Mega 6 Các bước để tiến hành phân tích và vẽ cây phát sinh chủng loại gồm có: 1. Xác định mục tiêu, lấy mẫu sinh vật và họ gene; Hình 9. Kết quả so sánh trình tự của mẫu với dữ liệu trên NCBI của phần mềm BLAST 27TẠP CHÍ NGHỀ CÁ SÔNG CỬU LONG - SỐ 8 - THÁNG 9/2016 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN II Hình 10. Màng hình chính cho Aligment của Mega 6 2. Chọn marker phân tử, tức gene hay protein cần đọc trình tự; 3. Đọc trình tự, hiệu chỉnh trình tự; 4. Sắp xếp thẳng hàng các trình tự; 5. Chọn mô hình tiến hóa; 6. Phân tích sự phát sinh chủng loài; 7. Đọc cây tiến hóa; 8. Kiểm tra cây tiến hóa; 9. Chấp nhận kết quả hoặc quay lại bước 2. Từ phầm mềm Bioedit, các bước từ 1 đến 4 sẽ được thực hiện, trong đó, các trình tự muốn nghiên cứu sẽ được gióng hàng và lưu trữ trên một file dữ liệu với đuôi được lưu là .fasta để chuẩn bị cho việc phân tích trong phần mềm Mega 6. Phần mềm Mega 6 là phần mềm phổ biến được sử dụng rộng rãi trong việc xử lý kết quả giải trình tự của các mẫu nghiên cứu. Sau đó, bước 5 và 6 sẽ được thực hiện với phần mềm Mega 6. Trong phần mềm này, cây phát sinh loài sẽ được thiết lập bằng chức nĕng Phylogeny (Hình 11). Trong Phylogeny, chọn phương pháp Test Neighbor-Joining Tree (NJ). Phương pháp neighbor-joining (NJ) có mô hình tiến hóa là một hàm hiện. Trong phương pháp này từng cặp trình tự một sẽ được so sánh thẳng hàng cặp đôi và ứng với từng cặp, khoảng cách di truyền sẽ được tính toán. Do mô hình tiến hóa là một hàm hiện nên một trong số mô hình tiến hóa có thể được chọn để tính toán khoảng cách di truyền giữa từng cặp taxa từ đó cho ra một ma trận khoảng cách giữa tất cả các taxa. Do phương pháp neighbor-joining mà một trong những phương pháp nhanh nhất để dò tìm cây tiến hóa nên nó thường được sử dụng để phân tích khối dữ liệu lớn với nhiều taxa. Đồng thời, trong phương pháp này sẽ chọn phân tích bootstrap 1000 lần lặp lại. Phân tích bootstrap được thực hiện nhằm kiểm tra tính chính xác và độ tin cậy cho từng nhánh trong cây tiến hóa. Đầu tiên, các vị trí từ chuỗi trình tự đã sắp xếp thẳng hàng sẽ được đảo chỗ cho nhau một cách ngẫu nhiên để tạo ra nhiều mẫu phụ (gọi là sự lặp lại bootstrap). Như vậy các mẫu phụ có kích 28 TẠP CHÍ NGHỀ CÁ SÔNG CỬU LONG - SỐ 8 - THÁNG 9/2016 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN II thước giống như mẫu gốc nhưng vị trí thành phần không giống nhau. Sau đó các mẫu phụ sẽ được trải qua quá trình phân tích tương tự như mẫu gốc đã trải qua. Kết quả từ những mẫu phụ sẽ được dùng để tính toán giá trị support (ủng hộ) cho một nhánh đơn lẻ nào đó. Do giá trị support được biểu diễn bằng tỷ lệ % nên ít nhất phải có 100 lần lặp lại, tức 100 lần tạo mẫu phụ. Thông thường thì người ta làm với 1.000 lần lặp lại để đạt được mức độ tin cậy cần thiết. Hình 11. Màn hình của phần mềm Phylogeny Dựa vào kết quả từ cây tiến hóa, chúng ta có thể xác định sự phát sinh chủng loại của các mẫu cần nghiên cứu dựa vào chỉ số được thể hiện trên từng nhánh cây. Ví dụ, theo hình 13, những mẫu cá dứa có mức độ khác biệt là 0 %, nhưng giữa các mẫu cá dứa và cá xác sọc là sự khác biệt này là 6,62 %. Điều này cho thấy hai loài này là hai loài khác nhau hoàn toàn do có độ chênh lệch hay khác biệt trên 2% (Hình 12). Hình 12: Cây Neighbor-Joining (NJ) được xây dựng từ trình tự gen Cytochrome b của 9 mẫu cá nghiên cứu và trình tự của 2 loài: Pangasius macronema và Pangasius elongatus 29TẠP CHÍ NGHỀ CÁ SÔNG CỬU LONG - SỐ 8 - THÁNG 9/2016 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN II Ngay sau khi có kết quả cây tiến hóa, tiến hành thực hiện bước 7 đến bước 9, nhà nghiên cứu có thể so sánh kết quả với cây tiến hóa mà nhà nghiên cứu đã định nghĩa sẵn từ trước. Sự sai khác giữa hai cây tiến hóa này có thể giúp nhà nghiên cứu đi đến quyết định chấp nhận kết quả hay quay lại hiệu chỉnh. Sau khi có cây tiến hóa, nhà nghiên cứu có thể đưa cây tiến hóa vào những chương trình xử lý hình ảnh đồ hòa thông thường để hiệu chỉnh hay làm nổi bật nhóm lòai cần chú ý trước khi công bố. Ngoài ra, Mega 6 cũng giúp tính toán khoảng cách di truyền của những loài nghiên cứu bằng chức nĕng Distance Compute pairwise distances dựa vào mô hình tính toán Kimura 2-Parameter (K2P). Kết quả nhận được như bảng 3, khoảng cách di truyền giữa các mẫu thuộc họ cá dứa và cá xác sọc là 0,137, kết quả này đã thể hiện sự khác biệt rõ rệt giữa hai loài này. Bảng 3: Khoảng cách di truyền của các mẫu thuộc 2 loài trong họ Pangasiidae: Pangasius macronema và Pangasius elongatus IV. KẾT LUẬN Quy trình phân tích đoạn gien Cytochrome b hoàn toàn phù hợp với mục đích nghiên cứu kiểm định cá Tra. Các mẫu DNA sau khi được thực hiện phản ứng PCR đã tinh sạch và giải trình tự. Các trình tự sau khi nhận từ công ty giải trình tự đã được kiểm tra chất lượng và xử lý bằng kết hợp hai phần mềm FinchTV và Bioedit để đạt được trình tự chuẩn xác nhất cho các bước phân tích tiếp theo. Sau đó, phần mềm BLAST của dữ liệu NCBI đã so sánh và xác định mức độ tương đồng của mẫu nghiên cứu với trình tự mẫu, nếu kết quả thể hiện là 99-100%, kết luận mẫu nghiên cứu có mức độ tương đồng cao với mẫu đã được công bố ở dữ liệu của NCBI. Cuối cùng, phần mềm Mega 6 sẽ là phần mềm thích hợp cho các phân tích về sự phát sinh loài và khoảng cách di truyền giữa cá Tra và những loài cá da trơn khác trong họ Pangasiidae. 30 TẠP CHÍ NGHỀ CÁ SÔNG CỬU LONG - SỐ 8 - THÁNG 9/2016 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN II TÀI LIỆU THAM KHẢO Hebert, P.D.N., Cywinska, A., Ball, S.L., deWaard, J.R., 2003. Biological identifications through DNA barcodes. Proceedings of the Royal Society of London. Series B: Biological Sciences 270, 313-321. Kimura, M., 1980. A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences. Journal of Molecular Evolution 16:111-120. MRC, 2008. Mekong River Commission, Phnom Penh Puckridge, M., Andreakis, N., Appleyard, S.A., Ward, R.D., 2013. Cryptic diversity in flathead fishes (Scorpaeniformes: Platycephalidae) across the Indo-West Pacific uncovered by DNA barcoding. Molecular Ecology Resources 13, 32-42. Tamura K, Dudley J, Nei M & Kumar S, 2007. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Molecular Biology and Evolution 24:1596- 1599. Sevilla R, Diez A, Noren M, 2007. Primers and polymerase chain reaction conditions for DNA barcoding teleost fish based on the mitochondrial cytochrome b and nuclear rhodopsin gienes. Mol Ecol Notes 7:730–734. Vences, M, 2005. Comparative performance of the 16S rRNA gene in DNA barcoding of of amphibians. (ngày 16/07/2016) 31TẠP CHÍ NGHỀ CÁ SÔNG CỬU LONG - SỐ 8 - THÁNG 9/2016 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN II PROCESS OF ANALYSIS FOR CYTOCHROME B GEN ON TRA CATFISH (Pangasianodon hypophthalmus) SAMPLES Tran Nguyen Ai Hang1*, Tran Thi Thuy Ha1 1 Department of Experimental Biology, Research Institute for Aquaculture No.2 2 Research Institute for Aquaculture No.1 *Email: hangtna.ria2@mard.gov.vn ABSTRACT For identify Tra catfish (Pangasianodonhypoph-thalmus), process of analysis for cytochrome b gen was built. Using the universal primer for cytochrome b gen to amplify PCR products about 430 bp for length from Tra catfish and other catfish samples belonging to Pangasidae. After cleaning process, products were sent to sequence. Before using for the analyzed steps, these sequences will be pre-treated to get the best sequences by two software such as FinchTV and Bioedit. Next, using BLAST to find a similarity with species was published in the database of NCBI. Finally, Mega 6 software would be use to analyze genetic distance as well as building Neighbor-Joining tree subspe- cies which also confirm close relationships between these species in the same family Pangasiidae. Keywords: Tra Catfish, Cytochrome b, Finch TV and Bioedit, Pangasianodon hypophthal- mus, Mega 6. Người phản biện: ThS. Bùi Thị Liên Hà Ngày nhận bài: 26/7/2016 Ngày thông qua phản biện: 10/8/2016 Ngày duyệt đĕng: 05/9/2016
File đính kèm:
 quy_trinh_phan_tich_doan_cytochrome_b_tren_cac_mau_ca_tra_pa.pdf
quy_trinh_phan_tich_doan_cytochrome_b_tren_cac_mau_ca_tra_pa.pdf

