Hiệu chỉnh các thành phần cho phản ứng PCR của các microsatellite và nghiên cứu đa dạng di truyền các quần thể cá tra (Pangasianodon hypophthalmus)
TÓM TẮT
Trong nghiên cứu này, các chỉ thị microsatellite khảo sát đặc hiệu cho bộ gen cho cá Tra
(Pangasianodon hypophthalmus) kế thừa từ các nghiên cứu trước đã được chọn lọc được để tối ưu
nhằm có thể tạo tổ hợp microsatellite có cùng điều kiện PCR tương đồng dùng trong quy trình
phản ứng multiplex PCR (một nhóm các microsatellite trong cùng một phản ứng PCR) phục vụ
phân tích đa dạng di truyền các quần thể cá Tra tại Việt Nam. Các chỉ thị sử dụng trong nghiên
cứu là bộ 7 chỉ thị microsatellite đơn (Ph07, Ph09, Ph41, CB12, CB15, CB18 và PSPG579) được
tối ứu phản ứng PCR thành công và thử nghiệm đánh giá đa dạng một số quần thể cá Tra nhằm
kiểm tra hiệu quả ứng dụng trong nghiên cứu tiếp theo. Phương pháp được sử dụng trong nghiên
cứu này là phản ứng PCR đơn khảo sát và tối ưu riêng cho từng chỉ thị microsatellite khi điện di
agarose 4% cho việc phân tích đa hình. Kết quả cho thấy tất cả chỉ thị đã được tối ưu thành công
cho các phản ứng PCR trên cá Tra và các chỉ thị có sự đa hình cao với trung bình số đoạn khuếch
đại 7,11, Na=6,19, He=0,78, PIC=0,77 trên mỗi chỉ thị trong 3 nhóm mẫu. Nghiên cứu làm nền
tảng cơ sở cho các bước khảo sát tiếp theo (Multiplex PCR) và có ý nghĩa khoa học trong trong
việc hỗ trợ các nghiên cứu đa dạng di truyền khác trên cá Tra.

Trang 1

Trang 2

Trang 3

Trang 4

Trang 5

Trang 6
Trang 7
Trang 8

Trang 9

Trang 10
Tải về để xem bản đầy đủ
Tóm tắt nội dung tài liệu: Hiệu chỉnh các thành phần cho phản ứng PCR của các microsatellite và nghiên cứu đa dạng di truyền các quần thể cá tra (Pangasianodon hypophthalmus)

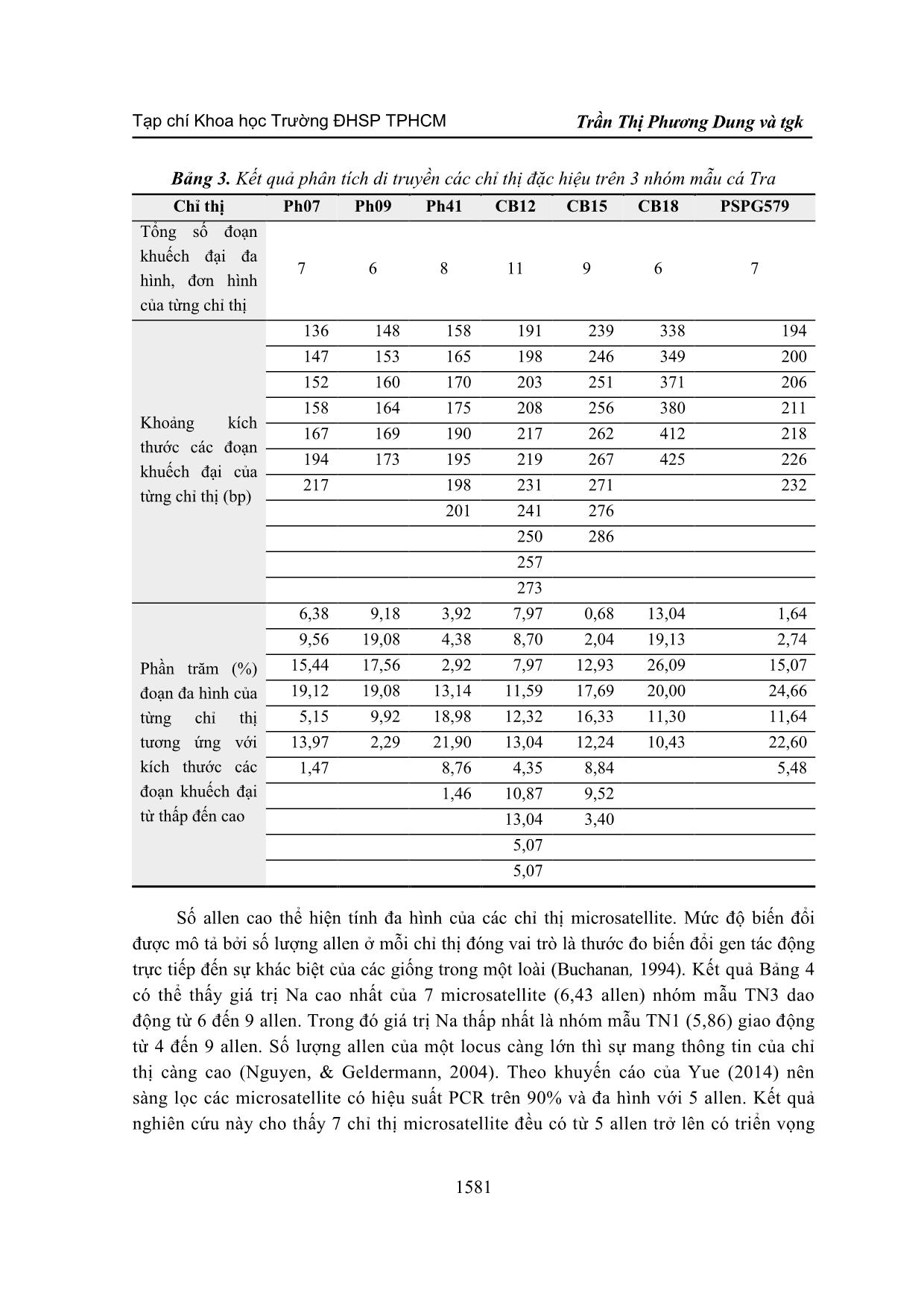
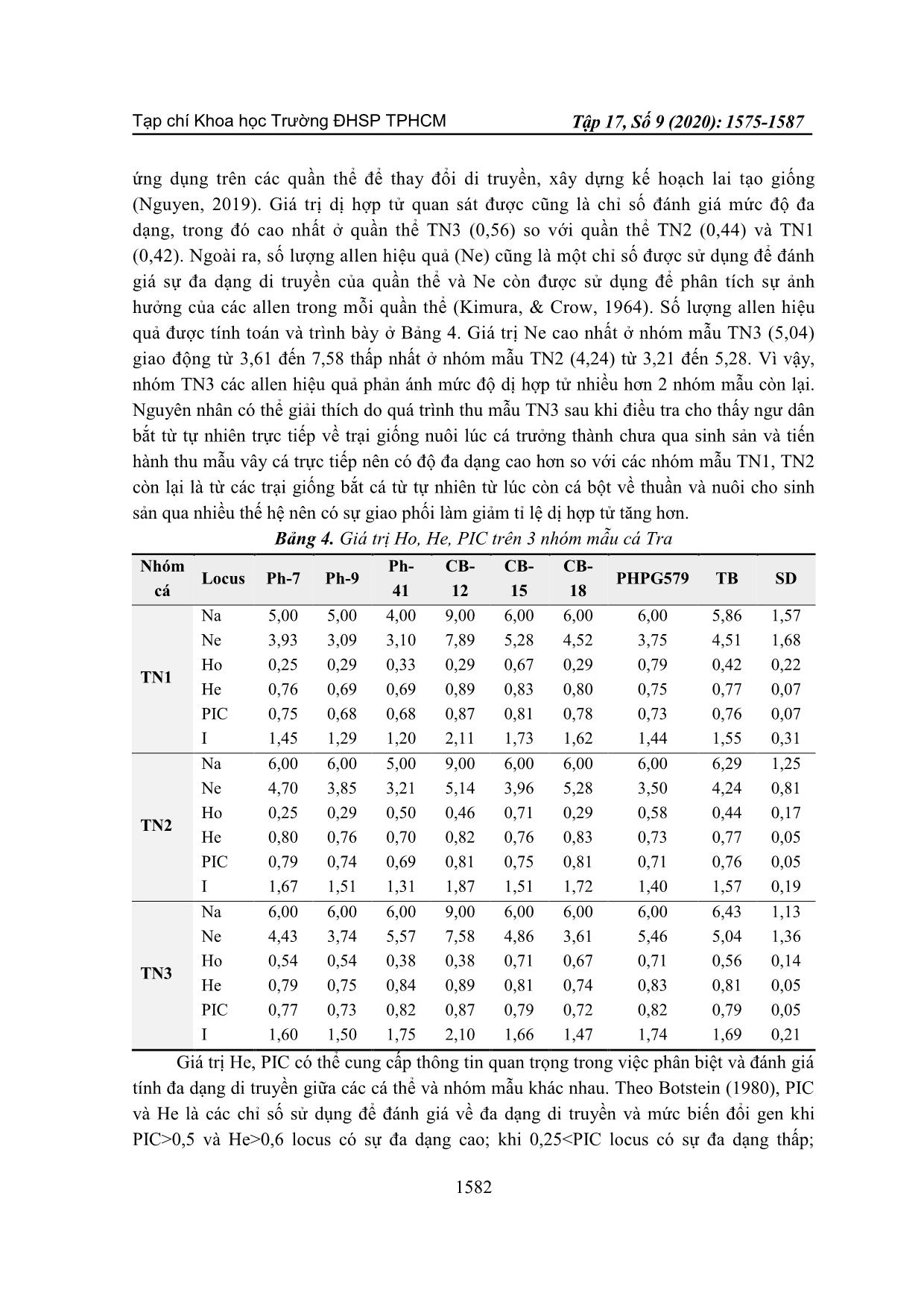
thấy ngư dân bắt từ tự nhiên trực tiếp về trại giống nuôi lúc cá trưởng thành chưa qua sinh sản và tiến hành thu mẫu vây cá trực tiếp nên có độ đa dạng cao hơn so với các nhóm mẫu TN1, TN2 còn lại là từ các trại giống bắt cá từ tự nhiên từ lúc còn cá bột về thuần và nuôi cho sinh sản qua nhiều thế hệ nên có sự giao phối làm giảm tỉ lệ dị hợp tử tăng hơn. Bảng 4. Giá trị Ho, He, PIC trên 3 nhóm mẫu cá Tra Nhóm cá Locus Ph-7 Ph-9 Ph- 41 CB- 12 CB- 15 CB- 18 PHPG579 TB SD TN1 Na 5,00 5,00 4,00 9,00 6,00 6,00 6,00 5,86 1,57 Ne 3,93 3,09 3,10 7,89 5,28 4,52 3,75 4,51 1,68 Ho 0,25 0,29 0,33 0,29 0,67 0,29 0,79 0,42 0,22 He 0,76 0,69 0,69 0,89 0,83 0,80 0,75 0,77 0,07 PIC 0,75 0,68 0,68 0,87 0,81 0,78 0,73 0,76 0,07 I 1,45 1,29 1,20 2,11 1,73 1,62 1,44 1,55 0,31 TN2 Na 6,00 6,00 5,00 9,00 6,00 6,00 6,00 6,29 1,25 Ne 4,70 3,85 3,21 5,14 3,96 5,28 3,50 4,24 0,81 Ho 0,25 0,29 0,50 0,46 0,71 0,29 0,58 0,44 0,17 He 0,80 0,76 0,70 0,82 0,76 0,83 0,73 0,77 0,05 PIC 0,79 0,74 0,69 0,81 0,75 0,81 0,71 0,76 0,05 I 1,67 1,51 1,31 1,87 1,51 1,72 1,40 1,57 0,19 TN3 Na 6,00 6,00 6,00 9,00 6,00 6,00 6,00 6,43 1,13 Ne 4,43 3,74 5,57 7,58 4,86 3,61 5,46 5,04 1,36 Ho 0,54 0,54 0,38 0,38 0,71 0,67 0,71 0,56 0,14 He 0,79 0,75 0,84 0,89 0,81 0,74 0,83 0,81 0,05 PIC 0,77 0,73 0,82 0,87 0,79 0,72 0,82 0,79 0,05 I 1,60 1,50 1,75 2,10 1,66 1,47 1,74 1,69 0,21 Giá trị He, PIC có thể cung cấp thông tin quan trọng trong việc phân biệt và đánh giá tính đa dạng di truyền giữa các cá thể và nhóm mẫu khác nhau. Theo Botstein (1980), PIC và He là các chỉ số sử dụng để đánh giá về đa dạng di truyền và mức biến đổi gen khi PIC>0,5 và He>0,6 locus có sự đa dạng cao; khi 0,25<PIC locus có sự đa dạng thấp; Tạp chí Khoa học Trường ĐHSP TPHCM Trần Thị Phương Dung và tgk 1583 0,25<PIC<0,5 locus có sự đa dạng trung bình. Kết quả ở Bảng 4 cho thấy trong số bốn nhóm mẫu, nhóm mẫu TN3 có chỉ số He và PIC cao nhất chứng tỏ nhóm mẫu TN3 có độ đa dạng di truyền cao hơn các nhóm mẫu còn lại. PIC trung bình của tất cả các chỉ thị và nhóm mẫu là 0,771. Hệ số PIC của hầu hết các microsatellite lớn hơn 0,5 và He của hầu hết các microsatellite lớn hơn 0,6 điều này chỉ ra rằng các chỉ thị microsatellite được chọn có độ đa dạng cao và có thể áp dụng cho phân tích đa dạng di truyền của các nhóm mẫu khác nhau ở cấp độ phân tử. Theo Berthouly (2008), các chỉ thị microsatellite được sử dụng để ước tính tính đa dạng kiểu gen của dị hợp tử và thông tin giá trị PIC trong việc lựa chọn và lai tạo các giống vật nuôi. Các chỉ thị microsatellite có đa hình cao được chọn để nghiên cứu các mối quan hệ di truyền và sự khác biệt về giống và có ý nghĩa trong sử dụng phân tích đa dạng di truyền các nhóm mẫu cá Tra tại Việt Nam. Hầu hết các chỉ thị đều cho thấy tỉ lệ dị hợp tử mong đợi He tương đối cao phản ánh sự tồn tại của sự khác biệt trong nhóm mẫu. Cây phân loài di truyền của các quần thể cá Tra được xây dựng dựa trên khoảng cách di truyền chuẩn của Nei (1972). Chương trình Dendrogam được xây dựng bằng phương pháp UPGMA của phần mềm NTSYSpc 2.11 cho kết quả khoảng cách di truyền gần nhất giữa nhóm mẫu TN1 và TN2, kế tiếp là TN3. Bốn nhóm mẫu được nhóm lại thành ba nhóm, nhóm gồm có TN1 và TN2, cùng chung nhóm với TN3 (Hình 3). Hình 3. Cây di truyền của 3 nhóm mẫu Khoảng cách di truyền nhỏ nhất được tìm thấy giữa TN1 và TN2 và cao nhất là giữa TN1 và TN3. Điều này cũng phù hợp với thực tế vì khi khảo sát các trại nuôi cá Tra trong nghiên cứu đều có nguồn gốc con giống xuất phát từ Campuchia. Kết quả này phù hợp với nghiên cứu của Tran (2017) cho thấy cá Tra tự nhiên nuôi tại các trại giống có khoảng cách di truyền thấp. Bước đầu các chỉ thị này có khả năng phục vụ phân tích đa dạng di truyền quần thể và phân tách di truyền giữa các quần thể. Tuy nhiên, cũng cần phải xem xét đến tính chưa ưu việt về kĩ thuật điện di đang sử dụng trong nghiên cứu này so với với các nghiên cứu khác (Nguyen, 2019) và những ảnh hưởng phụ có thể xảy ra trong quá trình khuếch đại sản phẩm cùng với phương thức điện di trên gel agarose như đã chỉ ra trong Tạp chí Khoa học Trường ĐHSP TPHCM Tập 17, Số 9 (2020): 1575-1587 1584 các nghiên cứu gần đây trên thế giới khi ứng dụng chỉ thị microsatellite (Harr, 2000; Brookes, 2012; Hosseinzadeh-Colagar, 2016). Vì thế, việc sử dụng các nguồn vật liệu di truyền từ nghiên cứu này là cơ sở cho những ứng dụng các công nghệ tiến bộ hơn trong phân tích chỉ thị là điều cần quan tâm (Nguyen, 2019). 4. Kết luận Bộ 7 chỉ thị microsatellite (CB12, CB15, CB18, Ph07, Ph09, Ph41 và PSP-G579) đã được thiết kế các thành phần cho phản ứng PCR và có thể được sử dụng để đánh giá đa dạng di truyền. Qua kết quả khảo sát nhận diện DNA thì có 7 chỉ thị khuếch đại được những đoạn DNA đa hình trong 3 nhóm mẫu với Na cao (>4 allen/quần thể) và hệ số PIC của hầu hết các microsatellite lớn hơn 0,5; He của hầu hết các microsatellite lớn hơn 0,6 điều này chỉ ra rằng các chỉ thị microsatellite được chọn có độ đa dạng cao và có thể áp dụng cho phân tích đa dạng di truyền của các nhóm mẫu. Ngoài ra, quy trình thử nghiệm đánh giá đa dạng di truyền trên cá Tra trong nghiên cứu này đã được tiến hành với một số kết quả nhất định như nhóm mẫu TN3 có độ đa dạng di truyền cao nhất so với hai nhóm mẫu còn lại. Khoảng cách di truyền giữa các nhóm cá Tra trong nghiên cứu nhóm 1 và nhóm 3 là xa nhất và khoảng cách di truyền giữa nhóm 1 và nhóm 2 là gần nhất. Kết quả nghiên cứu phần nào phản ánh mức độ đa dạng di truyền cũng như mối quan hệ của các quần thể cá Tra ở đồng bằng sông Cửu Long. Các chỉ thị microsatellite đều thể hiện tính đa hình bước đầu có ý nghĩa khi sử dụng trong đánh giá đa dạng di truyền nguồn vật liệu cá Tra và là cơ sở hỗ trợ các nghiên cứu khác trong nghiên cứu về di truyền phân tử trên cá Tra. Tuyên bố về quyền lợi: Các tác giả xác nhận hoàn toàn không có xung đột về quyền lợi. TÀI LIỆU THAM KHẢO Berthouly, C., Bed'Hom, B., Tixier-Boichard, M., Chen, C. F., Lee, Y. P., Laloë, D., Legros, H., Verrier, E., & Rognon, X. (2008). Using molecular markers and multivariate methods to study the genetic diversity of local European and Asian chicken breeds. Animal genetics, 39(2), 121-129. https://doi.org/10.1111/j.1365-2052.2008.01703.x Barker, J. S., Moore, S. S., Hetzel, D. J., Evans, D., Tan, S. G., & Byrne, K. (1997). Genetic diversity of Asian water buffalo (Bubalus bubalis): microsatellite variation and a comparison with protein-coding loci. Animal genetics, 28(2), 103-115. https://doi.org/10.1111/j.1365- 2052.1997.00085.x Botstein, D., White, R. L., Skolnick, M., & Davis, R. W. (1980). Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American journal of human genetics, 32(3), 314-331. Tạp chí Khoa học Trường ĐHSP TPHCM Trần Thị Phương Dung và tgk 1585 Buchanan, F. C., Galloway, S. M., & Crawford, A. M. (1994). Ovine microsatellites at the OarFCB5, OarFCB19, OarFCB20, OarFCB48, OarFCB129 and OarFCB226 loci. Animal genetics, 25(1), 60. Bui, T. L. H., Le, N. T. T., & Nguyen, V. S, (2017). Thu nghiem xac dinh pha he bang chi thi phan tu Microsatellite tren quan the chon giong ca Tra (Pangasianodon hypophthalmus) [Parentage assignment of selective population of striped catfish (Pangasianodon hypophthalmus) using microsatellite. Journal of Agriculture and Rural development, I, 8-97. Brookes, C., Bright, J. A., Harbison, S., & Buckleton, J. (2012). Characterising stutter in forensic STR multiplexes. Forensic science international. Genetics, 6(1), 58-63. https://doi.org/10.1016/j.fsigen.2011.02.001 Cruz, P., Ibarra, A. M., Mejia-Ruiz, H., Gaffney, P. M., & Pérez-Enríquez, R. (2004). Genetic variability assessed by microsatellites in a breeding program of Pacific white shrimp (Litopenaeus vannamei). Marine biotechnology (New York, N.Y.), 6(2), 157-164. https://doi.org/10.1007/s10126-003-0017-5 McDaniel, J., & Pillai, S. D. (2002). Gel alignment and band scoring for DNA fingerprinting using Adobe Photoshop. BioTechniques, 32(1), 120-123. https://doi.org/10.2144/02321bc03 Harr, B., Zangerl, B., & Schlötterer, C. (2000). Removal of microsatellite interruptions by DNA replication slippage: phylogenetic evidence from Drosophila. Molecular biology and evolution, 17(7), 1001-1009. https://doi.org/10.1093/oxfordjournals.molbev.a026381 Hogan, Z. S., & May, B. P. (2002). Twenty‐seven new microsatellites for the migratory Asian catfish family Pangasiidae. Molecular Ecology, 2, 38-41. doi:10.1046/j.1471- 8286.2002.00139.x Hosseinzadeh-Colagar, A., Haghighatnia, M. J., Amiri, Z., Mohadjerani, M., & Tafrihi, M. (2016). Microsatellite (SSR) amplification by PCR usually led to polymorphic bands: Evidence which shows replication slippage occurs in extend or nascent DNA strands. Molecular biology research communications, 5(3), 167-174. Kimura M., & Crow J. F., (1964). The number of alleles that can be maintained in a finite population. Genetic, 49(4), 725-738. Miah, G., Rafii, M. Y., Ismail, M. R., Puteh, A. B., Rahim, H. A., Islam, K., & Latif, M. A. (2013). A review of microsatellite markers and their applications in rice breeding programs to improve blast disease resistance. International journal of molecular sciences, 14(11), 22499-22528. https://doi.org/10.3390/ijms141122499 Miller, S. A., Dykes, D. D., & Polesky, H. F. (1988). A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic acids research, 16(3), 1215. https://doi.org/10.1093/nar/16.3.1215 Na-Nakorn, U., Sriphairoj, K., Sukmanomon, S., Poompuang, S., Kamonrat, W., (2006). Polymorphic microsatellite from DNA of the endangered Mekong giant catfish, Pangasianodon gigas (Chevey) and cross-species amplification in three species of Pangasius. Molecular Ecology, 6, 1174-1176. Nei, M. (1972). Genetic Distance between Populations. The American Naturalist, 106(949), 283- 292. Retrieved September 26, 2020, from Tạp chí Khoa học Trường ĐHSP TPHCM Tập 17, Số 9 (2020): 1575-1587 1586 Nguyen, T. D. T., & Geldermann H., (2004). Da dang di truyen mot so giong lon noi Viet Nam va Chau Au dua tren chi thi microsatellite [Genetic diversity of Vietnamese and European pig breeds based on microsatellite markers]. VNU Journal of Science, Natural Science and Technology, 20(4), 1-10. Nguyen, H. N., & Luu, T. H. G., (2012). Nghien cuu xac dinh da dang di truyen va huyet thong ca Tra (Pagasius hypophthalmus) bang chi thi microsatellite [Assessment of genetic diversity and parental assignment of striped catfish (Pagasius hypophthalmus) using microsatellite markers], Scientific report, Research Institute of Aquaculture No 1. Nguyen, N. T., Nguyen, T. K. N., Hoang, T. T., Vo, P. K. B. N., Phan, H. H. T., Nguyen, T. L. A. & Pham, C. T., (2019). Da dang di truyen mot so quan the trau noi Viet Nam [Genetic diversity of Vietnamese native buffaloes]. Journal of Animal Science and Technology, 242. Nguyen, T. P., & Nguyen, A. T., (2018). 50 nam nuoi ca Tra o dong bang song Cuu Long thanh cong va thach thuc trong phat trien ben vung [The success of 50-year striped catfish farming in the Mekong Delta and challeges on sustainable development]. Agricultural Pulishing House. Pham, A. T., & Nguyen, H. N., (2003). Nghien cuu bien di RFLPs mot so quan the ca Tra (Pangasius hypophthalmus) [Diversity of Tra catfish populations studied by RFLPs]. National Conference on Biotechnology, Hanoi. 724-726. So, N., & Vockaert, A. M., (2006), Genetic diversity and population history of the migratory catfishes Pangasianodon hypophthalmus and Pangasius bocourti in the Cambodian Mekong River. Fisheries science, 72, 469-476. Tran, N. H., & Phung, T. T., (2013). Danh gia da dang di truyen loai du sam da voi Keteleeria davidiana (Bertrand) Beissn bang ki thuat RAPD [Assessment of genetic diversity of Keteleeria davidiana (Bertrand) Beissn using RAPD technique]. Journal of Forest Science and Technology, (1), 22-27. Tran, T. T. H, Nguyen, T. H, Ngo, P. T., & Tran, N. A. H. (2017). Muc do da dang di truyen cua mot so quan the ca Tra su dung chi thi phan tu cytochrome b [Genetic diversity of Tra catfish populations in Vietnam using cytochrome b gene]. Journal of Science, Vinh University, 46(4A), 21-31 Volckaert, F. A. M., Hellemans, B., & Pouyaud, L., (1999). Nine polymorphic microsatellite markers in the SE Asian catfishes Pangasius hypophthalmus and Clarias batrachus. Animal Genetics, 30, 383-384. Yeh, F.C., Yang, R. C., Boyle, T. B. J., Ye, Z. H., & Mao, J. X. (1997). POPGENE, the User Friendly Shareware for Population Genetic Analysis. Molecular Biology and Biotechnology Centre, University of Alberta, Alberta. Yue, G. H., & Xia, J. H., (2014). Practical Considerations of Molecular Parentage Analysis in Fish. Journal of The World Aquaculture Society, 45(2), 89-103. Tạp chí Khoa học Trường ĐHSP TPHCM Trần Thị Phương Dung và tgk 1587 OPTIMAL RECTION PCR FOR RESEARCH GENETIC DIVERSITY OF TRA CATFISH (Pangasianodon hypophthalmus) Tran Thi Phuong Dung1*, Bui Thi Lien Ha2, Nguyen Hoang Thong2 Nguyen Ngoc Thuy Trang2, Tran Hoang Gia Linh2, Nguyen Van Sang2 1Ho Chi Minh City University of Education, Vietnam 2Research Institute for Aquaculture No. 2, Vietnam *Corresponding author: Tran Thi Phuong Dung – Email: dungttp@hcmue.edu.vn Received: March 26, 2020; Revised: April 28, 2020; Accepted: September 21, 2020 ABSTRACT In this experimental study, microsatellite markers for Tra catfish (Pangasianodon hypophthalmus) from previous studies were selected to optimize using the multiplex the process PCR to analyse the genetic diversity of Tra catfish populations in Vietnam. The markers used in the study is a set of seven single microsatellite markers (Ph07, Ph09, Ph41, CB12, CB15, CB18, and PSPG579) that have been optimized for successful PCR reactions in Tra catfish. The initial results of these markers were then applied to assess the genetic the diversity of Tra populations, showing that all microsatellite markers are polymorphic and meaningful when used to assess the genetic variation of Tra catfish populations. The method used in this study was single PCR and optimized for each microsatellite indicator with 4% agarose to analysis polymorphic’ populations. The results show that all indicators have been successfully optimized for PCR reactions on Tra catfish and the markers have a high polymorphism with an averaged amplification of 7.11, Na = 6.19, He = 0.78, and PIC = 0.77 on each marker in the three sample groups. The results will be served as a foundation for next steps on applying the multiplex PCR and support other studies on genetic diversity on Tra catfish. Keywords: microsatellite; PCR reaction; genetic diversity
File đính kèm:
 hieu_chinh_cac_thanh_phan_cho_phan_ung_pcr_cua_cac_microsate.pdf
hieu_chinh_cac_thanh_phan_cho_phan_ung_pcr_cua_cac_microsate.pdf

