Đặc điểm sinh hóa và di truyền của chủng Vibrio parahaemolyticus gây bệnh hoại tử gan thận cho cá mú nuôi tại Cát Bà, Hải Phòng
TÓM TẮT
Vi khuẩn Vibrio parahaemolyticus gây bệnh hoại tử gan thận cho nhiều loài cá biển có giá trị
kinh tế cao. Từ những mẫu cá mú mắc bệnh thu thập ở Cát Bà, Hải Phòng, đã phân lập được 6 mẫu
vi khuẩn có hình thái, đặc điểm sinh hóa đặc trưng cho chủng V. parahaemolyticus. 6 mẫu vi khuẩn
này đều có tính kháng với 5 loại kháng sinh nghiên cứu, đặc biệt có tính kháng cao với ampicillin.
Tỷ lệ sống sót của cá mú chấm cam (Epinephelus coioides) sau 14 ngày gây nhiễm với 6 mẫu vi
khuẩn là từ 2,2% đến 18,89% ở liều 100 μl/con, 107 CFU/ml. Khi phân tích sự có mặt của các gen
độc tố haemolysin, đã phát hiện được sự có mặt của 2 gen độc tố toxR và tlh trên cả 6 mẫu vi khuẩn.
Gen tdh và trh không được phát hiện trong hệ gen của 6 mẫu vi khuẩn phân lập. Đã khuếch đại được
trình tự gen toxR và tlh hoàn chỉnh với kích thước gen tương ứng lần lượt là 879 bp và 1254 bp với
hai cặp mồi toxR2- toxR4 và tlh1-tlh3 tự thiết kế. Kết quả nghiên cứu này là cơ sở cho những nghiên
cứu về đột biến gen, về cấu trúc không gian, cơ chế gây bệnh của những protein được mã hóa từ 2
gen độc tố này

Trang 1

Trang 2

Trang 3

Trang 4

Trang 5

Trang 6

Trang 7
Trang 8

Trang 9
Trang 10
Tải về để xem bản đầy đủ
Tóm tắt nội dung tài liệu: Đặc điểm sinh hóa và di truyền của chủng Vibrio parahaemolyticus gây bệnh hoại tử gan thận cho cá mú nuôi tại Cát Bà, Hải Phòng

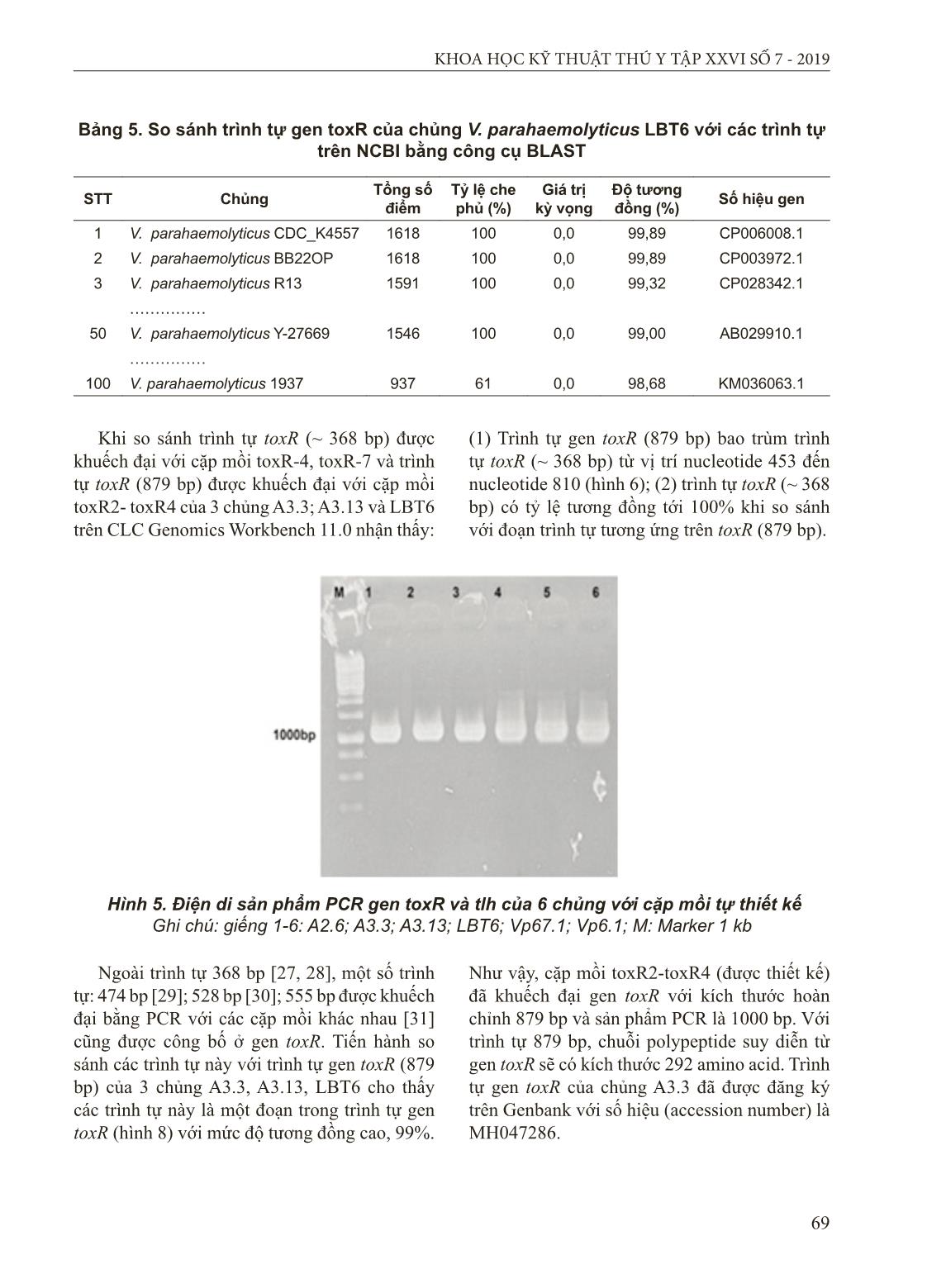
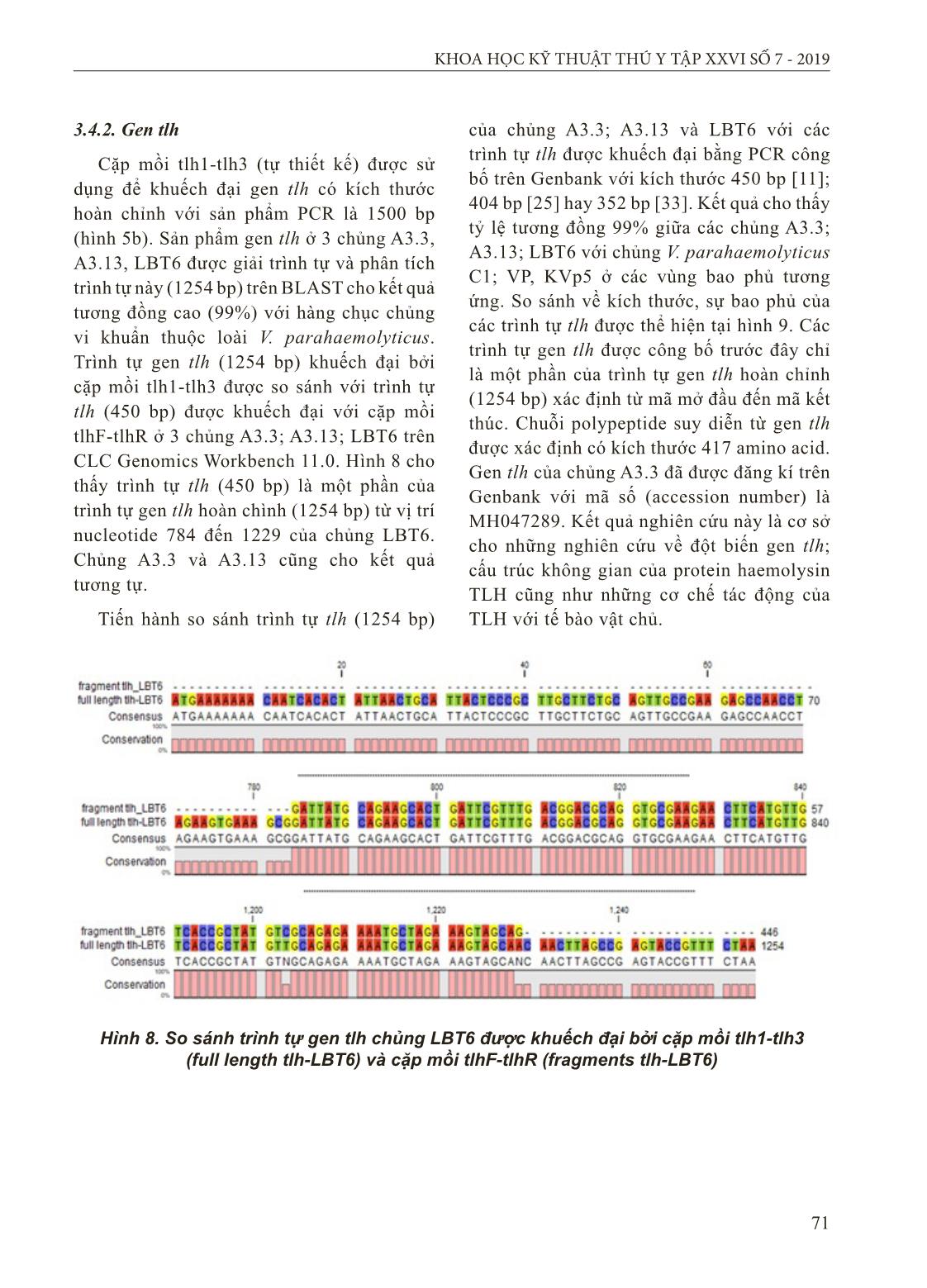
phân lập chứa gen trh và không phát hiện được gen tdh ở tất cả các mẫu vi khuẩn [26]. Tại Việt Nam, Nguyễn Văn Duy và cộng sự (2012) phân lập được 5 chủng vi khuẩn V. parahaemolyticus từ các mẫu hải sản tươi sống ở Nha Trang và đều không phát hiện được sự có mặt của hai gen tdh và trh trong hệ gen của chúng [27]. Như vậy, mặc dù chỉ có một gen mã hóa độc tố haemolysin (tlh) trong hệ gen, các chủng vi khuẩn phân lập trong nghiên cứu này vẫn có khả năng gây bệnh với tỷ lệ chết cao cho cá mú thí nghiệm. 3.4. Xác định kích thước gen toxR, tlh ở chủng Vibrio parahaemolyticus phân lập 3.4.1. Gen toxR Sử dụng cặp mồi toxR2-toxR4 (tự thiết kế) khuếch đại gen toxR cho sản phẩm có kích thước 1000 bp ở tất cả 6 chủng vi khuẩn (hình 5a). Sản phẩm này ở 3 chủng: A3.3; A3.13 và LBT6 được giải trình tự và phân tích bằng công cụ BLAST. Mức độ tương đồng của các trình tự này so với trình tự ở hàng chục chủng khác thuộc loài V. parahaemolyticus rất cao, 99% (bảng 5). Như vậy, cặp mồi toxR2-toxR4 được thiết kế là đặc hiệu, khuếch đại chính xác gen toxR của chủng V. parahaemolyticus. 69 KHOA HỌC KỸ THUẬT THÚ Y TẬP XXVI SỐ 7 - 2019 Khi so sánh trình tự toxR (~ 368 bp) được khuếch đại với cặp mồi toxR-4, toxR-7 và trình tự toxR (879 bp) được khuếch đại với cặp mồi toxR2- toxR4 của 3 chủng A3.3; A3.13 và LBT6 trên CLC Genomics Workbench 11.0 nhận thấy: (1) Trình tự gen toxR (879 bp) bao trùm trình tự toxR (~ 368 bp) từ vị trí nucleotide 453 đến nucleotide 810 (hình 6); (2) trình tự toxR (~ 368 bp) có tỷ lệ tương đồng tới 100% khi so sánh với đoạn trình tự tương ứng trên toxR (879 bp). Hình 5. Điện di sản phẩm PCR gen toxR và tlh của 6 chủng với cặp mồi tự thiết kế Ghi chú: giếng 1-6: A2.6; A3.3; A3.13; LBT6; Vp67.1; Vp6.1; M: Marker 1 kb Bảng 5. So sánh trình tự gen toxR của chủng V. parahaemolyticus LBT6 với các trình tự trên NCBI bằng công cụ BLAST STT Chủng Tổng số điểm Tỷ lệ che phủ (%) Giá trị kỳ vọng Độ tương đồng (%) Số hiệu gen 1 V. parahaemolyticus CDC_K4557 1618 100 0,0 99,89 CP006008.1 2 V. parahaemolyticus BB22OP 1618 100 0,0 99,89 CP003972.1 3 V. parahaemolyticus R13 1591 100 0,0 99,32 CP028342.1 50 V. parahaemolyticus Y-27669 1546 100 0,0 99,00 AB029910.1 100 V. parahaemolyticus 1937 937 61 0,0 98,68 KM036063.1 Ngoài trình tự 368 bp [27, 28], một số trình tự: 474 bp [29]; 528 bp [30]; 555 bp được khuếch đại bằng PCR với các cặp mồi khác nhau [31] cũng được công bố ở gen toxR. Tiến hành so sánh các trình tự này với trình tự gen toxR (879 bp) của 3 chủng A3.3, A3.13, LBT6 cho thấy các trình tự này là một đoạn trong trình tự gen toxR (hình 8) với mức độ tương đồng cao, 99%. Như vậy, cặp mồi toxR2-toxR4 (được thiết kế) đã khuếch đại gen toxR với kích thước hoàn chỉnh 879 bp và sản phẩm PCR là 1000 bp. Với trình tự 879 bp, chuỗi polypeptide suy diễn từ gen toxR sẽ có kích thước 292 amino acid. Trình tự gen toxR của chủng A3.3 đã được đăng ký trên Genbank với số hiệu (accession number) là MH047286. 70 KHOA HỌC KỸ THUẬT THÚ Y TẬP XXVI SỐ 7 - 2019 Hình 6. So sánh trình tự gen toxR chủng LBT6 được khuếch đại bởi 2 cặp mồi toxR2-toxR4 (full length toxR-LBT6) và toxR-4, toxR-7 (fragments toxR-LBT6) Hình 7. So sánh chiều dài các đoạn trình tự gen toxR (màu xám) khuếch đại bằng PCR ở các chủng vi khuẩn V. parahaemolyticus (A) Gen toxR hoàn chỉnh của chủng A3.3/A3.13/LBT6 được khuếch đại bởi cặp mồi toxR2- toxR4; (B) đoạn gen toxR và toxS của chủng KP34 với số hiệu DQ845170.1 [32] và các đoạn gen toxR của (C) chủng CECT611 với số hiệu FM202714.1 [31], (D) chủng ATCC 17802 với số hiệu GQ228073.1 [29], (E) chủng C1 với số hiệu JQ929913.1 [27] và chủng A3.3/A3.13/LBT6 được khuếch đại bởi cặp mồi toxR-4, toxR-7. 71 KHOA HỌC KỸ THUẬT THÚ Y TẬP XXVI SỐ 7 - 2019 3.4.2. Gen tlh Cặp mồi tlh1-tlh3 (tự thiết kế) được sử dụng để khuếch đại gen tlh có kích thước hoàn chỉnh với sản phẩm PCR là 1500 bp (hình 5b). Sản phẩm gen tlh ở 3 chủng A3.3, A3.13, LBT6 được giải trình tự và phân tích trình tự này (1254 bp) trên BLAST cho kết quả tương đồng cao (99%) với hàng chục chủng vi khuẩn thuộc loài V. parahaemolyticus. Trình tự gen tlh (1254 bp) khuếch đại bởi cặp mồi tlh1-tlh3 được so sánh với trình tự tlh (450 bp) được khuếch đại với cặp mồi tlhF-tlhR ở 3 chủng A3.3; A3.13; LBT6 trên CLC Genomics Workbench 11.0. Hình 8 cho thấy trình tự tlh (450 bp) là một phần của trình tự gen tlh hoàn chình (1254 bp) từ vị trí nucleotide 784 đến 1229 của chủng LBT6. Chủng A3.3 và A3.13 cũng cho kết quả tương tự. Tiến hành so sánh trình tự tlh (1254 bp) của chủng A3.3; A3.13 và LBT6 với các trình tự tlh được khuếch đại bằng PCR công bố trên Genbank với kích thước 450 bp [11]; 404 bp [25] hay 352 bp [33]. Kết quả cho thấy tỷ lệ tương đồng 99% giữa các chủng A3.3; A3.13; LBT6 với chủng V. parahaemolyticus C1; VP, KVp5 ở các vùng bao phủ tương ứng. So sánh về kích thước, sự bao phủ của các trình tự tlh được thể hiện tại hình 9. Các trình tự gen tlh được công bố trước đây chỉ là một phần của trình tự gen tlh hoàn chỉnh (1254 bp) xác định từ mã mở đầu đến mã kết thúc. Chuỗi polypeptide suy diễn từ gen tlh được xác định có kích thước 417 amino acid. Gen tlh của chủng A3.3 đã được đăng kí trên Genbank với mã số (accession number) là MH047289. Kết quả nghiên cứu này là cơ sở cho những nghiên cứu về đột biến gen tlh; cấu trúc không gian của protein haemolysin TLH cũng như những cơ chế tác động của TLH với tế bào vật chủ. Hình 8. So sánh trình tự gen tlh chủng LBT6 được khuếch đại bởi cặp mồi tlh1-tlh3 (full length tlh-LBT6) và cặp mồi tlhF-tlhR (fragments tlh-LBT6) 72 KHOA HỌC KỸ THUẬT THÚ Y TẬP XXVI SỐ 7 - 2019 IV. KẾT LUẬN Từ các mẫu cá mú nghi mắc bệnh hoại tử gan thận ở Cát Bà, Hải Phòng phân lập được 6 chủng vi khuẩn có hình thái, đặc điểm phù hợp với V. parahaemolyticus. Các vi khuẩn này đều là vi khuẩn gram âm, có khả năng lên men glucose, không lên men lactose, không sinh H 2 S, không sinh khí, sinh indol, sinh catalase, có khả năng di động và làm tan huyết dạng β. Các chủng vi khuẩn này đều có khả năng gây bệnh trở lại cho cá mú chấm cam với tỷ lệ sống sót của cá từ 2,22% đến 18,89%. Phát hiện được 2 gen độc tố (toxR và tlh) trong hệ gen của 6 chủng vi khuẩn. Hai gen này đã được khuếch đại với trình tự hoàn chỉnh 879 bp (gen toxR) và 1254 bp (gen tlh) với 2 cặp mồi tự thiết kế (toxR2-toxR4; tlh1-tlh3). Lời cảm ơn: Công trình được hoàn thành với kinh phí đề tài trọng điểm cấp Viện Đại học Mở Hà Nội (mã số: V2018.01-01) và đề tài cấp trường Đại học Sư phạm Hà Nội (mã số: SPHN 18-03). TÀI LIỆU THAM KHẢO 1. Mohamad N, Mustafa M, Amal MNA, Saad MZ, Md Yasin IS, Al-Saari N. Environmental Factors Associated with the Presence of Vibrionaceae in Tropical Cage-Cultured Marine Fishes. J Aquat Anim Health 2019; 31(2):154-67. 2. Nguyễn Thị Thanh Thùy, Nguyễn Hữu Dũng, I.Wergeland H. Thành phần protein và độc tính của vi khuẩn Vibrio parahaemolyticus gây bệnh lở loét trên cá mú chấm cam (Epinephelus coioides) nuôi thương phẩm tại Khánh Hòa. Tạp chí khoa học - Công nghệ thủy sản 2012; 4/2012:180-4. 3. Nguyễn Bá Hiên. Giáo trình miễn dịch học ứng dụng; NXB Đại học Nông nghiệp Hà Nội; 2010. 4. Nguyễn Trọng Nghĩa, Âu Thị Kim Ngọc, Nguyễn Thị Minh Trang, Đặng Thị Hoàng Oanh, Trương Quốc Phú, Phạm Anh Tuấn. Phân lập và xác định khả năng gây hoại tử gan tụy của vi khuẩn Vibrio parahaemolyticus. Hội nghị khoa học trẻ ngành thủy sản toàn quốc lần thứ IV 2013; Kỷ yếu:411-9. 5. Nishibuchi M, JB K. Thermostable direct hemolysin gene of Vibrio parahaemolyticus a virulence gene acquyred by a marine bacterium. Infection and immunity 1995; 63(6):2093-9. 6. Li L, Gao M, Lu T, Gu D. Dissection of ToxR-dependent and ToxR-independent stress-regulated pathways in Vibrio parahaemolyticus. Microbiological Research 2019; 223- 225:79-87. 7. Zhou S, Gao ZX, Zhang M, Liu DY, Zhao XP, Liu Y. Development of a quadruplex loop-mediated isothermal amplification assay for field detection of four Vibrio species associated with fish disease. Springerplus 2016; 5:1104. 8. Klein SL, Gutierrez West CK, Mejia DM, Lovell CR. Genes similar to the Vibrio parahaemolyticus virulence-related genes tdh, tlh, and vscC2 occur in other vibrionaceae species isolated from a pristine estuary. Appl Environ Microbiol 2014; 80:595-602. Hình 9. Sơ đồ biểu diễn của các đoạn gen tlh do PCR khuếch đại (màu xám) ở các chủng vi khuẩn V. parahaemolyticus (A) gen tlh hoàn chỉnh của chủng A3.3/LBT6/A2.6 được khuếch đại bởi cặp mồi tlh1-tlh3 và đoạn gen tlh của (B) chủng C1 với accession number JQ929914.1 [27] và chủng A3.3/A2.6/ LBT6 khuếch đại bởi cặp mồi tlhF-tlhR (C) chủng VP với accession number AY829372.1 [25] (D) chủng KVp5 với accession number HM195239.1 [33]. 73 KHOA HỌC KỸ THUẬT THÚ Y TẬP XXVI SỐ 7 - 2019 9. Zulkifli Y, Alitheen NB, Son R, Yeap SK, Lesley MB, Raha AR. Identification of Vibrio parahaemolyticus isolates by PCR targeted to the toxR gene and detection of virulence genes. International Food Research Journal 2009; 16:289-96. 10. Subhashini N, Krishnaiah N, C BK. Detection of Vibrio parahaemolyticus in shell fish by cultrural and polymerase chain reaction. International Journal of Pharma and Bio Sciences 2011; 2:335-41. 11. Yáñez R, Bastías R, G. H, Salgado O, Katharios P, Romero J, et al., Amplification of tlh gene in other Vibrionaceae specie by specie-specific multiplex PCR of Vibrio parahaemolyticus. Electronic Journal of Biotechnology 2015; 18:459-63. 12. Bej AK, Patterson DP, Brasher CW, Vickery MC, Jones DD, CA K. Detection of total and hemolysin-producing Vibrio parahaemolyticus in shellfish using multiplex PCR amplification of tlh, tdh and trh., J Microbiol Methods 1999; 36(3):215-25. 13. Kim S, Chung HY, Lee DH, Lim JG, Kim SK, Ku HJ, et al., Complete genome sequence of Vibrio parahaemolyticus strain FORC_008, a foodborne pathogen from a flounder fish in South Korea. Pathog Dis 2016; 74(5):ftw044. 14. Trần Linh Phước. Phương pháp phân tích vi sinh vật trong nước, thực phẩm và mỹ phẩm; Nxb Giáo dục; 2009. 15. Institure. CaLS, Performance standards for antimicrobial disk and dilution susceptibility tests of bacteria isolate from aquatic animals, In: Third Edition M-A, editor., Clinical and Laboratory Standards Institure, Wayne, JR.: Approve Standard; (2006a). 16. Paranjpye RN, Myers MS, Yount EC, Thompson JL. Zebrafish as a model for Vibrio parahaemolyticus virulence. Microbiology 2013; 159:2605-15. 17. Kim YB, Okuda J, Matsumoto C, Takahashi N, Hashimoto S, M N. Identification of Vibrio parahaemolyticus strains at the species level by PCR targeted to the toxR gene., J Clin Microbiol 1999; 37(4):1173-7. 18. Honda T, Iida T. The pathogenicity of Vibrio parahaemolyticus and the role of the thermostable direct haemolysin and related haemolysins. Reviews in Medical Microbiology 1993; 4:106-13. 19. Zulkifli Y, Alitheen NB, Son R, Raha AR, Yeap SK, M. N. Antibiotic resistance and plasmid profiling of Vibrio parahaemolyticus isolated from cockles in Padang, Indonesia. International Food Research Journal 2009; 16:53-8 20. Xu X, Cheng J, Wu Q, Zhang J, Xie T. Prevalence, characterization, and antibiotic susceptibility of Vibrio parahaemolyticus isolated from retail aquatic products in North China. BMC Microbiol 2016; 16:32. 21. Shyne APS, Sobbhana KS, George KC, RR P. Phenotypic characteristics and antibiotic sensitivity of Vibrio parahaemolyticus strains isolated from diseases grouper (Epinephelus spp.). J Mar Biol Ass 2008; 50:1-6. 22. Li J, Yie J, Foo RWT, Ling JML, Xu HS, Woo NYS. Antibiotic resistance and plasmid profiles of vibrio isolates from cultured silver sea bream, Sparus sarba. Marine Pollution Bulletin 1999; 39:245-9. 23. Iida T, Suthienkul O, Park KS, Tang GQ, Yamamoto RK, Ishibashi M, et al., Evidence for genetic linkage between the ure and trh genes in Vibrio parahaemolyticus. J Med Microbiol 1997; 46:639-45. 24. Nishibuchi M, Kaper JB. Thermostable direct hemolysin gene of Vibrio parahaemolyticus: a virulence gene acquyred by a marine bacterium. Infection and immunity 1995; 63:2093-9. 25. Xie ZY, Hu CQ, Chen C, Zhang LP, Ren CH. Investigation of seven Vibrio virulence genes among Vibrio alginolyticus and Vibrio parahaemolyticus strains from the coastal mariculture systems in Guangdong, China. Lett Appl Microbiol 2005; 41:202-7. 26. Chakraborty RD, Surendran PK. Occurrence and distribution of virulent strains of Vibrio parahaemolyticus in seafoods marketed from Cochin (India). World Journal of Microbiology and Biotechnology 2008; 24:1929-35. 27. Nguyễn Văn Duy, Nguyễn Thị Cẩm Ly. Phân lập và xác định gen độc tố của Vibrio parahaemolyticus trong hải sản tươi sống ở Nha Trang. Tạp chí khoa học - Công nghệ thủy sản 2012; 2/2012:42-7. 28. Wong HC, Chen CH, Chung YJ, Liu SH, Wang TK, Lee CL, et al., Characterization of new O3:K6 strains and phylogenetically related strains of Vibrio parahaemolyticus isolated in Taiwan and other countries. J Appl Microbiol 2005; 98:572-80. 29. Wang D, Yu S, Chen W, Zhang D, Shi X. Enumeration of Vibrio parahaemolyticus in oyster tissues following artificial contamination and depuration. Lett Appl Microbiol 2010; 51:104-8. 30. Hoffmann M, Monday SR, Fischer M, Brown EW. Genetic and phylogenetic evidence for misidentification of Vibrio species within the Harveyi clade. Lett Appl Microbiol 2012; 54:160-5. 31. Pascual J, Macian MC, Arahal DR, Garay E, Pujalte MJ. Multilocus sequence analysis of the central clade of the genus Vibrio by using the 16S rRNA, recA, pyrH, rpoD, gyrB, rctB and toxR genes. Int J Syst Evol Microbiol 2010; 60:154-65. 32. Vongxay K, Pan Z, Zhang X, Wang S, Cheng S, Mei L, et al., Occurrence of pandemic clones of Vibrio parahaemolyticus isolates from seafood and clinical samples in a Chinese coastal province. Foodborne Pathog Dis 2008; 5(2):127-34. 33. Collin B, AS. R-H. Occurrence and potential pathogenesis of Vibrio cholerae, Vibrio parahaemolyticus and Vibrio vulnificus on the South Coast of Sweden. FEMS Microbiol Ecol 2011; 78(2):306-13. Ngày nhận 13-8-2019 Ngày phản biện 23-8-2019 Ngày đăng 1-11-2019
File đính kèm:
 dac_diem_sinh_hoa_va_di_truyen_cua_chung_vibrio_parahaemolyt.pdf
dac_diem_sinh_hoa_va_di_truyen_cua_chung_vibrio_parahaemolyt.pdf

