Đặc trưng di truyền của chủng IHHNV phân lập tôm nuôi ở đồng bằng sông Cửu Long
TÓM TẮT
Sự đa dạng di truyền của chủng IHHNV đã được xác định trên nhiều vùng địa lý khác nhau trên
thế giới. Trong nghiên cứu này, khi phân tích và giải trình tự gen IHHNV trên những mẫu tôm sú
nuôi ở Đồng bằng sông Cửu Long (ĐBSCL) cho thấy đã có sự hiện diện chủng virus IHHN type A
không lây bệnh, đoạn gen khuếch đại 1100 bp của chủng này có độ tương đồng rất cao với đoạn gen
chủng IHHNV type A phân lập ở Úc và Mandagasca là 100%. Đồng thời, chủng virus IHHN type
lây nhiễm cũng đã được xác định hiện diện trên tôm sú nuôi vùng này. Phân tích trình tự cho thấy
chủng có kí hiệu KG phân lập tại tôm nuôi ở Kiên Giang và chủng kí hiệu ST phân lập trên tôm
con ở Bình Thuận nhưng nuôi Sóc Trăng mất đi một đoạn khoảng 79 nucleotide, tuy nhiên sự mất
này không ảnh hưởng đến các vùng đọc mở của bộ gen để mã hóa protein của virus. Phân tích trình
tự của chủng IHHNV KG có sự tương đồng rất cao với các chủng IHHNV ở Hawaii, Mexico, Đài
Loan C và Ecuador, độ tương đồng bộ gen trên 95%. Và một chủng IHHNV type lây nhiễm khác
có kí hiệu ST, phân tích trình tự chủng này cho thấy có độ tương đồng thấp đối với chủng có kí hiệu
KG và chủng IHHNV phân lập ở Hawaai, nhưng có độ tương đồng cao với chủng IHHNV phân lập
ở Đài Loan A, B và Thái Lan. Trình tự bộ gen của 2 chủng KG và ST có kích thước khoảng 3824
bp. Kết quả nghiên cứu cho thấy, tôm sú nuôi ở ĐBSCL có sự hiện diện 2 chủng IHHNV type lây
nhiễm và chủng IHHNV type A không lây nhiễm

Trang 1

Trang 2

Trang 3

Trang 4

Trang 5

Trang 6

Trang 7

Trang 8
Trang 9

Trang 10
Tải về để xem bản đầy đủ
Tóm tắt nội dung tài liệu: Đặc trưng di truyền của chủng IHHNV phân lập tôm nuôi ở đồng bằng sông Cửu Long

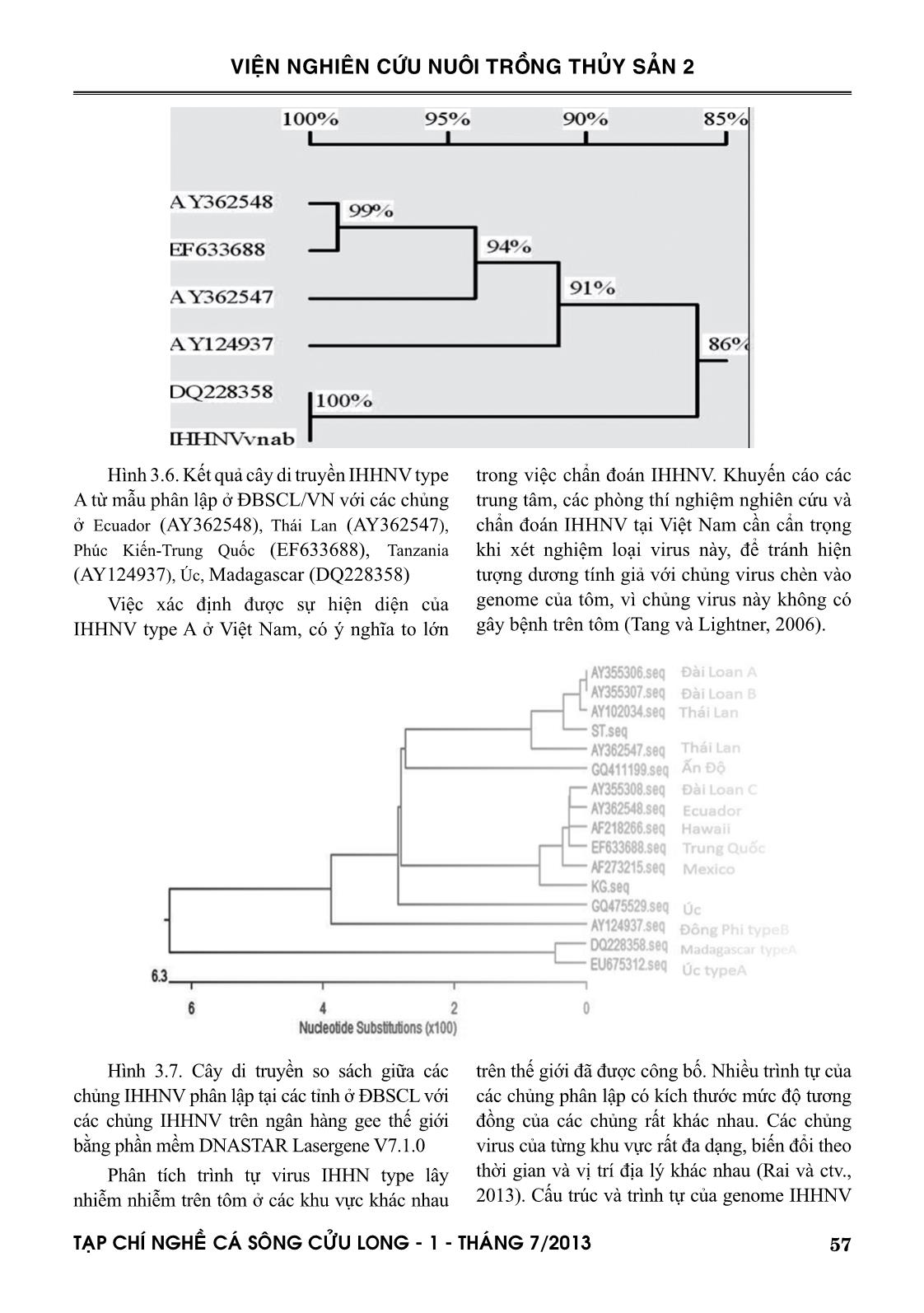
N Trình tự gen IHHNV type A/B nhiễm trên tôm sú nuôi đã được nhiều nhà nghiên cứu phân tích và giải trình tự (Tang và ctv., 2006). Trên cơ sở đó chúng tôi tiến hành so sánh trình tự và mối quan hệ di truyền của IHHNV type A phân lập ở tôm sú nuôi ở ĐBSCL với trình tự IHHNV type lây nhiễm và type A/B ở các khu vực như type lây nhiễm của Ecuador (AY362548), Thái Lan (AY362547), Phúc Kiến-Trung Quốc (EF633688), type B của Tanzania (AY124937) và type A của Úc, Madagascar (DQ228358). Sau khi so sánh trình tự IHHNV type A được phân lập từ ĐBSCL với trình tự IHHNV của các nước: Ecuador, Thái Lan, Phúc Kiến-Trung Quốc, Tanzania và Úc, Madagascar cho thấy trình tự IHHNV type A của Việt Nam có sự tương đồng khá cao so với trình tự gen IHHNV type A ở Úc, Madagascar (DQ228358) do Tang và ctv công bố (2006). Độ tương đồng cao nhất với trình tự IHHNV type A của Việt Nam là 100% với chủng IHHNV type A phân lập ở Úc. Sự tương đồng cao này có thể do IHHNV type A của Việt Nam có nguồn gốc từ Úc, Châu phi (Madagascar) vì tôm bố mẹ của Việt Nam được nhập từ nhiều nước trong đó có Úc và Châu Phi. Trái lại thì trình tự IHHNV type A của Việt Nam có độ tương đồng thấp với trình tự IHHNV type B của Tanzania AY124937 chỉ 86%. Ngoài ra chúng tôi thiết lập cây di truyền so sánh trình tự IHHNV type A tại Việt nam với các khu vực trên ngân hàng gen như hình 3.6. Bảng 2: Trình tự tương đồng nucleotide trên bộ gen của các chủng phân lập ở ĐBSCL với một số chủng ở các nước khác trên thế giới. 57TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 1 - THAÙNG 7/2013 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN 2 Hình 3.6. Kết quả cây di truyền IHHNV type A từ mẫu phân lập ở ĐBSCL/VN với các chủng ở Ecuador (AY362548), Thái Lan (AY362547), Phúc Kiến-Trung Quốc (EF633688), Tanzania (AY124937), Úc, Madagascar (DQ228358) Việc xác định được sự hiện diện của IHHNV type A ở Việt Nam, có ý nghĩa to lớn trong việc chẩn đoán IHHNV. Khuyến cáo các trung tâm, các phòng thí nghiệm nghiên cứu và chẩn đoán IHHNV tại Việt Nam cần cẩn trọng khi xét nghiệm loại virus này, để tránh hiện tượng dương tính giả với chủng virus chèn vào genome của tôm, vì chủng virus này không có gây bệnh trên tôm (Tang và Lightner, 2006). Hình 3.7. Cây di truyền so sách giữa các chủng IHHNV phân lập tại các tỉnh ở ĐBSCL với các chủng IHHNV trên ngân hàng gee thế giới bằng phần mềm DNASTAR Lasergene V7.1.0 Phân tích trình tự virus IHHN type lây nhiễm nhiễm trên tôm ở các khu vực khác nhau trên thế giới đã được công bố. Nhiều trình tự của các chủng phân lập có kích thước mức độ tương đồng của các chủng rất khác nhau. Các chủng virus của từng khu vực rất đa dạng, biến đổi theo thời gian và vị trí địa lý khác nhau (Rai và ctv., 2013). Cấu trúc và trình tự của genome IHHNV 58 TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 1 - THAÙNG 7/2013 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN 2 kí hiệu ST so với các chủng IHHNV phân lập ở Thái Lan và Đài Loan có độ tương đồng cao lên đến 99,3%. Tuy nhiên chủng IHHNV có kí KG có trình tự lại tương đồng cao với chủng IHHNV phân lập ở Hawaii trên 98%, so với chủng IHHNV Mexico, Đài Loan C và Ecuador trên 95%. Theo các nghiên cứu trước, Rai và ctv (2011), hầu hết các chủng IHHNV lây nhiễm đều có 3 khung đọc mở mã hóa cho protein ORF (open reading frame): ORF1 mã hóa cho protein giả định 1 (NS1) có kích thước khoảng 2001 bp, đoạn ORF2 mã hóa cho protein giả định (NS2) có kích thước khoảng 1092 bp và ORF3 mã hóa cho protein vỏ virus (VP) có kích thước khoảng 990 bp. Ở chủng IHHNV có kí hiệu ST và KG đều có 3 vùng đọc mở mã hóa protein (OFR1, OFR2 và OFR3). ORF1 ở vị trí 570-2570 có kích thước là 2001 bp mã hóa cho protein NS1, vùng mã hóa cho protein NS2 là ORF2 ở vị trí 514- 1605 nucleotide có kích thước là 1092 bp, và mã hóa cho protein vỏ là ORF3 có vị trí là 2512-501 nucleotide có kích thước là 990 bp. So sánh trình tự của đoạn ORF1 của chủng kí hiệu KG với các chủng khác thì độ tương đồng cao nhất với chủng IHHNV ở Hawaii, Đài Loan A, Đài Loan C và Ecuador (99,5%), tỷ lệ tương đồng thấp hơn đối với chủng IHHNV của Madagasca (87,2%), Tanzania (92,3%) và Úc (GQ411199, 94,8%), tỷ lệ tương đồng so với chủng IHHNV phân lập tại ĐBSCL và Ấn Độ (≥ 96,3%). Kết quả này cũng tương đương với trình tự amino aicd, tương đồng cao nhất với chủng ở Hawaii (98,8%), Đài Loan A, C và Ecuador (98,5%). Mặc dù tỷ lệ nucleotide của chủng Ấn Độ và chủng này có tỷ lệ trung bình cao (96,3%), nhưng trình tự amino acid tương đồng rất thấp (91,3%), ngược lại chủng IHHNV phân lập tại Úc có trình tự nucleotide tương đồng thấp (94,8%) có trình tự amino acid cao (95,9%). Kết quả này cho thấy, chủng này có sự khác biệt về độc lực do sự biến đổi các trình tự nucleotide dẫn đến sự khác biệt về sự mã hóa amino acid trong cấu trúc của virus. Đặc biệt, sự khác biệt rõ rệt là vị trí đọc mở ORF1 giữa 2 chủng phân lập trên tôm sú ở ĐBSCL, ở chủng kí hiệu KG đoạn OFR1 mã hóa amino acid (ATG) ở vị trí 514 nt của gen và đoạn OFR1 mã hóa amino acid (ATG) ở vị trí 570 nt đối với chủng kí hiệu ST. Nếu đột biến nhiều sẽ dẫn đế sự thay đổi tổng hợp các amino acid và dẫn đến hiện tượng đột biến chủng và tính độc lực sẽ biến đổi theo (Tang và Lightner., 2002). Vì vậy sự khác biệt này có thể dẫn đến độc lực và sự lây nhiễm của hai chủng IHHNV có kí hiệu ST và KG khác nhau. Khi so sánh trình tự bộ gen chủng IHHNV kí hiệu KG với các chủng IHHNV khác trên thế giới cho thấy độ tương đồng cao nhất đối với chủng phân lập ở Hawaii, Mỹ (AF28266) lên đến 98,8 %, kế đến là chủng phân lập Mexico (AF273215), Trung Quốc (EF633688) là 98,6% và xa với chủng Úc type A (86,1%), Tanzania type A là 91,7%. Bảng 3: So sánh trình tự nucleotide của đoạn mã hóa cho protein của virus IHHN phân lập tại ĐBSCL với các chủng trên thế giới 59TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 1 - THAÙNG 7/2013 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN 2 Chủng virus IHHN phân lập có kí hiệu ST, kích thước khoảng 3824 bp. Chủng này tương đồng rất cao, với A+T của toàn bộ gen là 56,82% tương tự như virus IHHN phân lập Thái Lan (AY102034) 56,9 %. Chủng IHHNV phân lập có kí hiệu ST có trình tự tương đồng rất cao với chủng IHHNV của Thái Lan (AY102034), Đài Loan A (AY355306). Chủng IHHNV phân lập có kí hiệu ST có trình tự ORF1 tương đồng rất cao với chủng IHHNV của Thái Lan (AY102034) 99,8%, Đài Loan B (AY355307) 99,8% và chủng IHHNV phân lập ở Kiên Giang có kí hiệu KG 96,5%. ORF1 của chủng này mã hóa cho protein giả định NS1 có vị trí là (570-2570 nucleotide), chiều dài đoạn protein khoảng 666 amino acid với mã khởi động ATG ở vị trí 570 nucleotide của bộ gen và mã kết thúc TAA ở vị trí 2570, có trọng lượng phân tử là 75,8 kDa. Đoạn ORF2 có vị trí (514-1605 nt) mã hóa cho protein NS2 với khoảng 363 amino acid có trọng lượng phân tử 42,1 kDa, trình tự của vùng mã hóa này cũng tương đồng rất cao với các chủng Thái Lan 2000, Đài Loan B (99,9%). ORF3 mã hóa protein vỏ của virus có vị trí trên bộ gen (2512- 3501 nucleotide) với chiều dài là 990 bp mã hóa cho protein này 329 amino acid. Trình tự tương đồng của đoạn ORF3 so với các chủng trên thế giới Đài Loan B, Thái Lan (99,5%). Kết quả trên cho thấy chủng phân lập IHHNV kí hiệu TG và chủng phân lập ST có thể là cùng 1 chủng và chủng này có họ hàng gần với chủng phân lập ở Đài Loan B, Thái Lan 2000. V. KẾT LUẬN Trong nghiên cứu này, chúng tôi đã xác định chủng virus IHHN type A hiện diện ở Việt Nam, trình tự chủng phân lập khi phân tích một đoạn gen cho thấy chủng này có độ tương đồng rất cao với chủng IHHNV type A ở Madagascar và Úc (100%). Phân lập được 2 bộ gen của chủng IHHNV type lây nhiễm trên tôm sú nuôi ở các tỉnh ở ĐBSCL và thành công trong việc giải hoàn toàn trình tự bộ gen của các chủng virus này. Phân tích trình tự cho thấy chủng IHHNV phân lập có kí hiệu ST có độ tương đồng rất cao lên đến 99,7% và có họ hàng gần với chủng IHHNV phân lập ở Đài Loan A, B và Thái Lan. Chủng IHHNV phân lập có kí hiệu KG có trình tự và họ hàng gần với chủng ở Hawaii, Mexico, Đài Loan C và Ecuador, độ tương đồng bộ gen trên 95%.Trong nghiên cứu này đã khẳng định được sự hiện diện IHHNV type A gắn vào genome tôm sú và sự hiện diện IHHNV type lây nhiễm (1 và 2) trên tôm sú nuôi ở ĐBSCL. Trình tự genome của hai chủng IHHNV phân lập ở ĐBSCL có kí hiệu ST và KG đã được đăng kí trên ngân hàng gen với mã số KC513422 và JX84006. TÀI LIỆU THAM KHẢO Bonami JR, Trumper B, Mari J, Brehelin M, Lightner DV, 1990. Purification and characterization of the infectious hypodermal and hematopoietic necrosis virus of penaeid shrimps. J Gen Virol 71, 2657-2664. Flegel TW, 2006. Detection of major penaeid shrimp viruses in Asia, a historical perspective with emphasis on Thailand. Aquaculture 258, 1-33. Krabsetsve K, Cullen BR, Owens L, 2004. Rediscovery of the Australian strain of infectious hypodermal and haematopoietic necrosis virus. Dis. Aquat. Organ 61, 153-158. Lightner DV, Redman RM, and Bell TA, 1983. Infectious hypodermal and hematopoietic necrosis, a newly recognized virus disease of penaeid shrimp. J. Invertebr. Pathol. 42, 62-70. Nunan L, Poulos BT, Lightner DV, 2000. Use of polymerase chain reaction for the detection of infectious hypodermal and hematopoietic necrosis virus in penaeid shrimp. Mar. Biotechnol. 2 (4), 319-328. OIE, 2009. Diagnostic Manual for Aquatic Animal Diseases 6th Edition. Office International des Epizooties (OIE), Paris. Chapter 2.2.2. Infectious Hypodermal and Hematopoietic Necrosis, pp. 78- 95. Phạm Đặng Thiên An, Phạm Văn Hùng, Hoàng Hiếu Ngọc, Phạm Hùng Vân, 2009. Giải trình tự bộ gen infectious hypodermal and hematopoietic necrosis virus gây bệnh hoại tử trên tôm. Y học thành phố Hồ Chí Minh, 13 (2), 129-137. Rai P, Safeena M.P, Karunasagar I, Karunasagar I, 60 TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 1 - THAÙNG 7/2013 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN 2 2011. Complete nucleic acid sequence of Penaeus stylirostris densovirus (PstDNV) from India. Virus Research 158, 37-45. Rai P, Safeena MP, Karunasagar I, Karunasagar I, 2009. Simultaneous presence of infectious hypodermal and hematopoietic necrosis virus (IHHNV) and Type A virus-related sequence in Penaeus monodon from India. Aquaculture, 295, 168-174. Rai P, Pradeep B, Safeena MP., Krabsetsve K, La Fauce K, Owens L and Karunasagar I, 2013. Genomics, molecular epidemiology and diagnostics of infectious hypodermal and hematopoietic necrosis virus. Indian Journal of Virology, 23 (2), pp. 203-214. Saksmerprome V, Puiprom O, Noonin C, Flegel TW, 2010. Detection of infectious hypodermal and haematopoietic necrosis virus (IHHNV) in farmed Australian Penaeus monodon by PCR analysis and DNA sequencing. Aquaculture 298, 190-193. Shike H, Dhar AK, Burns JC, Shimizu C, Jousset FX, Klimpel KR, Bergoin M, 2000. Infectious hypodermal and hematopoietic necrosis virus of shrimp is related to mosquito brevidensoviruses. Virology 277, 167-177. Sukhumsirichart W, Attasart P, Boonsaeng V, Panyim S., 2006. Complete nucleotide sequence and genomic organization of hepatopancreatic parvovirus (HPV) of Penaeus monodon. Virology 346, 266 - 277. Tang KFJ and Lightner DV, 2002. Low sequence variation among isolates of infectious hypodermal and hematopoietic necrosis virus (IHHNV) originating from Hawaii and the Americas. Dis Aquat Org 49, 93-97. Tang KFJ and Lightner DV, 2006. Infectious hypodermal and hernatopoietic necrosis virus (IHHNV)-related sequences in the genome of the black tiger prawn Penaeus monodon from Africa and Australia. Virus Research, 118, 185-191. Tang KFJ, Navarro SA and Lightner DV, 2007. A PCR assay for discriminating between infectious hypodermal and hematopoietic necrosis virus (IHHNV) and the virus-related sequences in the genome of Penaeus monodon. Dis. Aquat. Org. 74, 165-170. Tang KFJ, Poulos BT, Wang J, Redman RM, Shih HH, Lightner DV, 2003. Geographic variations among infectious hypodermal and hematopoietic necrosis virus (IHHNV) isolates and characteristics of their infection. Dis. Aquat. Organ. 53, 91-99. Website: SEQUENCE AND CHARACTERISTIC OF INFECTIOUS HYPODERMAL AND HEMATOPOIETIC NECROSIS VIRUS (IHHNV) ISOLATED FROM THE CULTURED BLACK TIGER SHRIMP (Peneaus monondon) IN MEKONG DELTA Cao Thanh Trung1, Nguyen Thị Kim My2 and Pham Hung Van3 ABSTRACT Genetic IHHNV genome of variants has been charateristed in different geographical areas in the word. In this study, analyzing and sequencing of IHHNV were isolated in the black tiger shrim in Mekong Delta. The type A IHHNV-related sequence has been detected in cultured P. monodon in Mekong Delta. An amplified 1200 fragment of type A IHHNV-related sequence is highest identity with type A IHHNV-related sequence isolated shrimp from Australia and Madagasca (100%). For type IHHNV infection, the infectious IHHNV strain isolated from the shrimps in Kieng Giang province was designated IHHNV-KG and the strain isolated from postlarve shrimp in the Soc Trang was designated IHHNV-TS. Used overlapping PCR primers to amplify all of the IHHNV genome 61TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 1 - THAÙNG 7/2013 VIỆN NGHIÊN CỨU NUÔI TRỒNG THỦY SẢN 2 from the Mekong Delta samples to sequence were analized. Comparison of IHHNV-KG sequence with GenBank records of IHHNV isolates. Comparing the 2 genomes of IHHNV infected in P. monodon in Mekong Delta with the genome of IHHNV of Hawaii that were lost a 79 nucleotides. However, it did not impact to open reading frame (ORFs) coding protein of virus. The IHHNV-KG strain was found that it had more 95% identity to sequences of infectious IHHNV from American, Mexico, Taiwan C and Ecuador. Other infectious IHHNV-ST, it had a low similar sequence with IHHNV-KG, but it had a high sequence identity with the infectious IHHNV from Taiwan A, B and Thailand. The size genome of two IHHNV strains isolated from Mekong Delta is 3825 bp. In this study, the IHHNV-related sequence and infectious IHHNV were detected in the cultured P. mon- odon in Mekong Delta. Key words: Mekong Delta, Peneus monodon, Infectious hypodermal and hematopoietic necrosis virus (IHHNV) Người phản biện: ThS. Nguyễn Thị Hiền Ngày nhận bài: 6/6/2013 Ngày thông qua phản biện: 6/7/2013 Ngày duyệt đăng: 8/7/2013 1 Southern Monitoring Center for Aquaculture Environment & Epidemic - Research Institute for Aquaculture No.2 Email: thanhtrung77@yahoo.com 2 Faculty of Biology – Ho Chi Minh city University of Science, 227 Nguyen Van Cu st, District 5, Ho Chi Minh City, Vietnam 3 Nam khoa Biotech Company, 793/58 Tran Xuan Soan st - Tân Hưng ward, distric 7, Ho Chi Minh City, Vietnam
File đính kèm:
 dac_trung_di_truyen_cua_chung_ihhnv_phan_lap_tom_nuoi_o_dong.pdf
dac_trung_di_truyen_cua_chung_ihhnv_phan_lap_tom_nuoi_o_dong.pdf

