Ứng dụng công nghệ sinh học trong chẩn đoán ký sinh trùng truyền lây qua cá
TÓM TẮT
Công nghệ sinh học được ứng dụng nhiều trong nuôi trồng thủy sản nói chung và trong
chẩn đoán tác nhân gây bệnh thủy sản nói riêng. Bài viết này giới thiệu ứng dụng công
nghệ sinh học trong chẩn đoán ký sinh trùng truyền lây qua cá, cụ thể là sán lá ruột nhỏ
Haplorchis thường ký sinh ở ruột non của người và động vật ăn cá nhiễm ấu trùng sán
chưa được xử lý triệt để. Do trứng, ấu trùng và sán trưởng thành có kích thước nhỏ,
hình dạng giống một số loài sán lá khác nên dễ chẩn đoán và nhận dạng nhầm. Một
phương pháp sinh học phân tử lần đầu tiên được áp dụng ở Việt Nam nhằm xác định
sán lá ruột nhỏ bao gồm H. taichui và H. pumilio dựa trên phản ứng PCR gen ITS-2 với
mồi xuôi là 3SF: 5’-GGTACCGGTGGATCACTCGGCTCGTG-3’ và mồi ngược BD2R:
5’-TATGCTTAAATTCAGCGGGT-3’), sử dụng chung cho các loài sán lá ở nhiệt độ
bám mồi là 50oC, và giải trình trình tự phân tích gen. Nguồn mẫu được dùng cho nghiên
cứu gồm sán trưởng thành ký sinh trên người và ấu trùng metacercaria ký sinh trên cá
được thu ở Nam Định (Việt Nam) và Bangkok (Thái Lan). Chuỗi gen ITS-2 thu được có
độ dài 290 bp là của H. pumilio và 446 bp là của H. taichui. Trình tự sắp xếp nucleotide
trong gen ITS-2 của H. taichui chênh lệch so với H. pumilio do một đoạn gen có độ dài
154 nucleotide được chèn vào từ nucleotide thứ 198 đến 352. Có sự tương đồng cao (99-
100%) trong gen ITS-2 giữa mẫu sán Việt Nam và mẫu sán Thái Lan trong nghiên cứu.

Trang 1
Trang 2

Trang 3
Trang 4

Trang 5

Trang 6

Trang 7

Trang 8

Trang 9
Tóm tắt nội dung tài liệu: Ứng dụng công nghệ sinh học trong chẩn đoán ký sinh trùng truyền lây qua cá

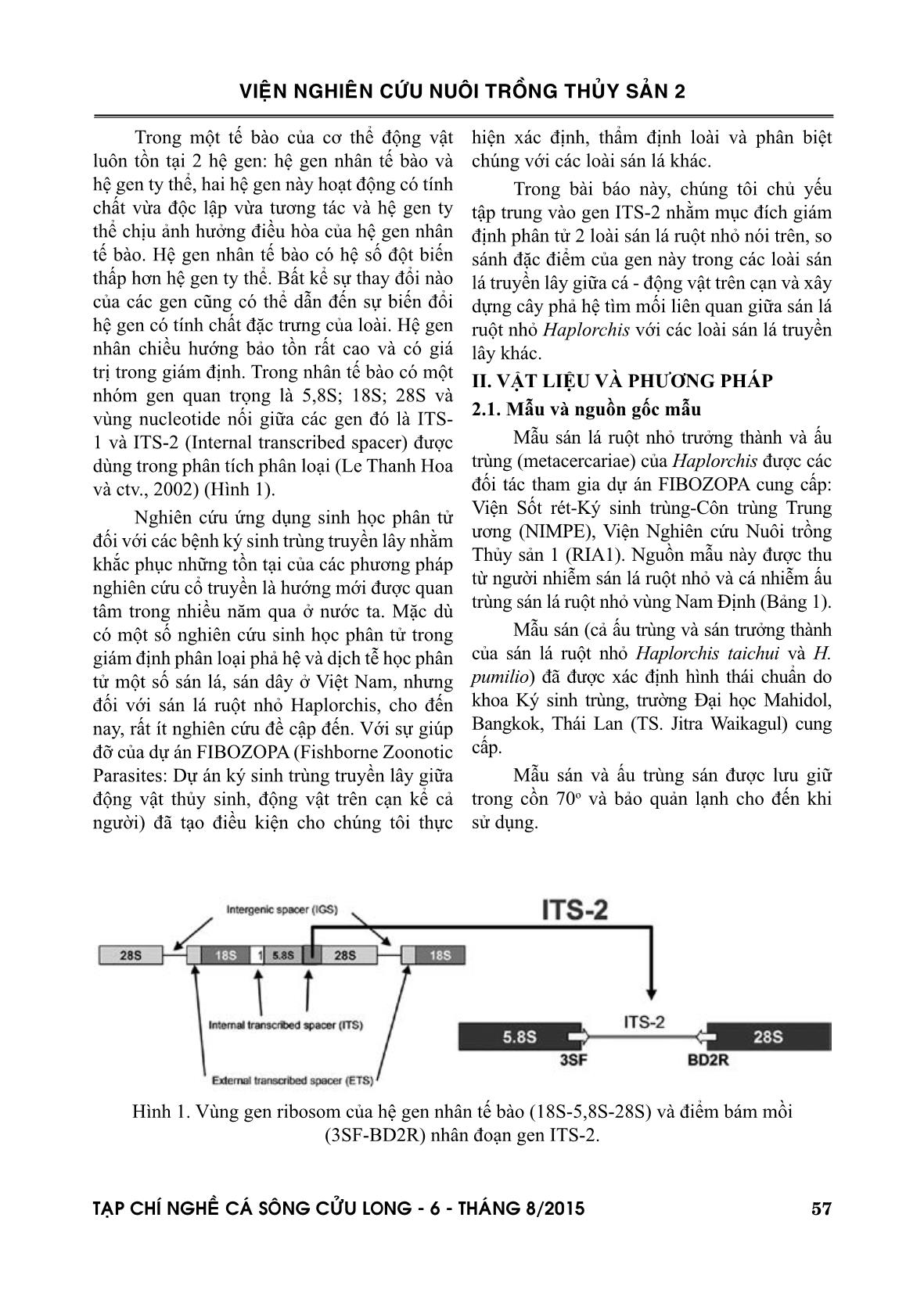
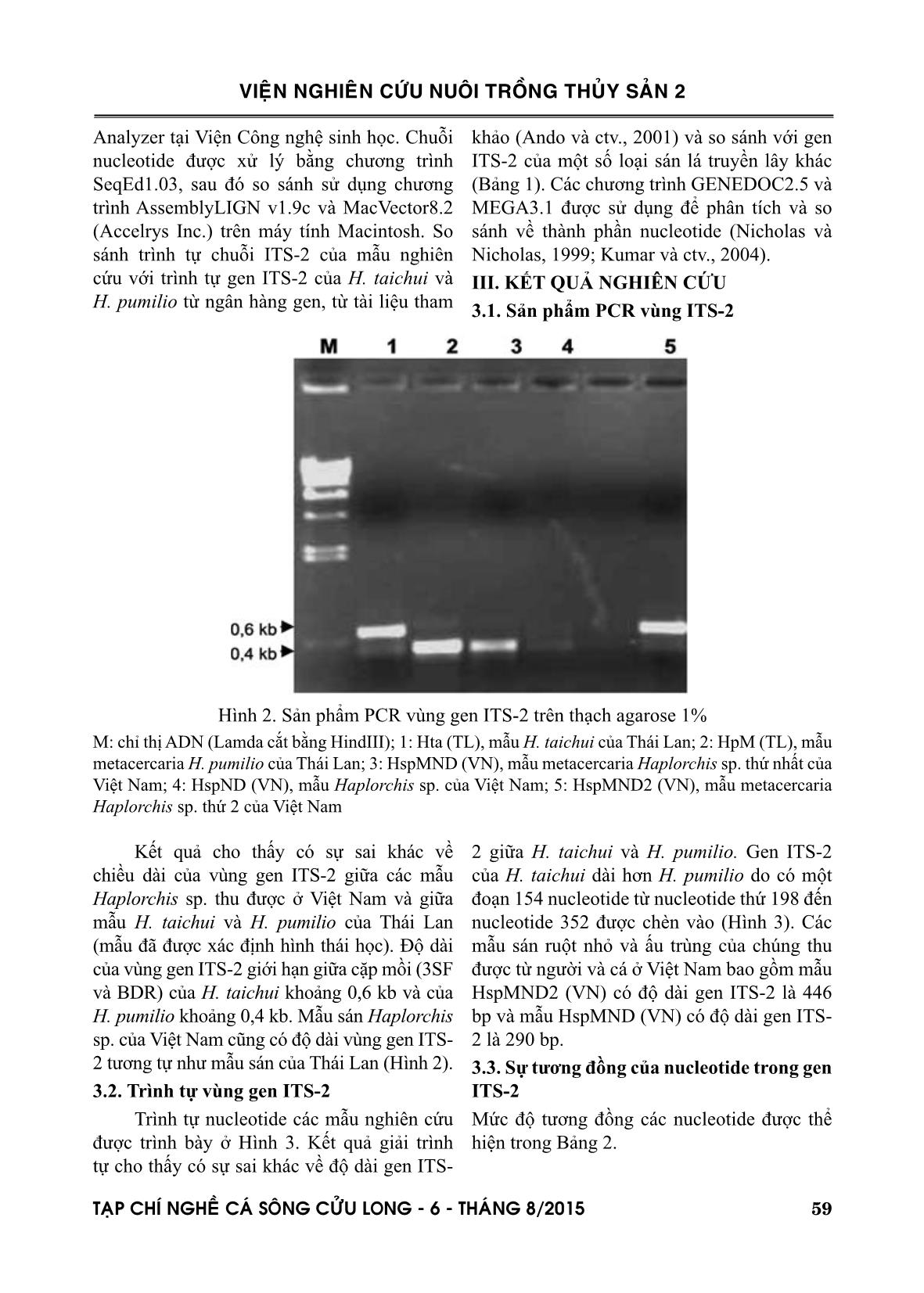
100 Avant Genetic Loài sán Ký hiệu mẫu Loại mẫu (ADN) Nguồn mẫu Ký chủ Nguồn gen ITS-2 H. pumilio Hpu (TL1) Sán trưởng thành Thái Lan Người Ando và ctv., 2001 H. pumilio HpM (TL) Metacercaria Thái Lan Cá Nghiên cứu này Haplorchis sp. HspMND (VN) Metacercaria Việt Nam Cá Nghiên cứu này H. pumilio Hpu (AY245706) Dzikowski và ctv., 2004; AY245706 Haplorchis sp. HspND(VN) Sán trưởng thành Việt Nam Người Nghiên cứu này Haplorchis sp. HspNDV(VN) Sán trưởng thành Việt Nam Người Nghiên cứu này H. taichui Hta(TL1) Sán trưởng thành Thái Lan Người Ando và ctv., 2001 Haplorchis sp. HspMND2(VN) Metacercaria Việt Nam Cá Nghiên cứu này H. taichui Hta(AY245705) Dzikowski và ctv., 2004; AY245705 O. viverrini Ovi(AY584735) Sanath và ctv., 2004; AY584735 O. viverrini Ovi(TL1) Sán + ấu trùng Thái Lan Người Ando và ctv., 2001 C. sinensis Csi(SKR) Lee và ctv., 1999; AF217095 Bảng 1. Danh sách mẫu, nguồn gốc của mẫu trong nghiên cứu và nguồn gen tham khảo 2.2. Phân tích ITS-2 Quá trình phân tích sinh học phân tử được thực hiện tại Phòng thí nghiệm của Phòng Miễn dịch học, Viện Công nghệ Sinh học, Viện Khoa học và Công nghệ Việt Nam (Hà Nội). Mẫu sán lá ruột nhỏ trưởng thành và ấu trùng sán được tách chiết ADN tổng số bằng QIAamp DNA kit (QIAGEN Inc., USA). Sau khi thu được ADN tổng số tiến hành thực hiện phản ứng PCR với chu trình nhiệt: Nhiệt độ để bung liên kết là 94oC trong 1 phút, nhiệt độ bám mồi là 50oC trong 1 phút, nhiệt độ cung cấp cho quá trình tổng hợp chuỗi ADN là 72oC trong vòng 2 phút. Toàn bộ quá trình thực hiện phản ứng PCR là 35 chu kỳ (Le Thanh Hoa và ctv., 2002). VIEÄN NGHIEÂN CÖÙU NUOÂI TROÀNG THUÛY SAÛN 2 TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 6 - THAÙNG 8/2015 59 khảo (Ando và ctv., 2001) và so sánh với gen ITS-2 của một số loại sán lá truyền lây khác (Bảng 1). Các chương trình GENEDOC2.5 và MEGA3.1 được sử dụng để phân tích và so sánh về thành phần nucleotide (Nicholas và Nicholas, 1999; Kumar và ctv., 2004). III. KẾT QUẢ NGHIÊN CỨU 3.1. Sản phẩm PCR vùng ITS-2 Analyzer tại Viện Công nghệ sinh học. Chuỗi nucleotide được xử lý bằng chương trình SeqEd1.03, sau đó so sánh sử dụng chương trình AssemblyLIGN v1.9c và MacVector8.2 (Accelrys Inc.) trên máy tính Macintosh. So sánh trình tự chuỗi ITS-2 của mẫu nghiên cứu với trình tự gen ITS-2 của H. taichui và H. pumilio từ ngân hàng gen, từ tài liệu tham Hình 2. Sản phẩm PCR vùng gen ITS-2 trên thạch agarose 1% M: chỉ thị ADN (Lamda cắt bằng HindIII); 1: Hta (TL), mẫu H. taichui của Thái Lan; 2: HpM (TL), mẫu metacercaria H. pumilio của Thái Lan; 3: HspMND (VN), mẫu metacercaria Haplorchis sp. thứ nhất của Việt Nam; 4: HspND (VN), mẫu Haplorchis sp. của Việt Nam; 5: HspMND2 (VN), mẫu metacercaria Haplorchis sp. thứ 2 của Việt Nam Kết quả cho thấy có sự sai khác về chiều dài của vùng gen ITS-2 giữa các mẫu Haplorchis sp. thu được ở Việt Nam và giữa mẫu H. taichui và H. pumilio của Thái Lan (mẫu đã được xác định hình thái học). Độ dài của vùng gen ITS-2 giới hạn giữa cặp mồi (3SF và BDR) của H. taichui khoảng 0,6 kb và của H. pumilio khoảng 0,4 kb. Mẫu sán Haplorchis sp. của Việt Nam cũng có độ dài vùng gen ITS- 2 tương tự như mẫu sán của Thái Lan (Hình 2). 3.2. Trình tự vùng gen ITS-2 Trình tự nucleotide các mẫu nghiên cứu được trình bày ở Hình 3. Kết quả giải trình tự cho thấy có sự sai khác về độ dài gen ITS- 2 giữa H. taichui và H. pumilio. Gen ITS-2 của H. taichui dài hơn H. pumilio do có một đoạn 154 nucleotide từ nucleotide thứ 198 đến nucleotide 352 được chèn vào (Hình 3). Các mẫu sán ruột nhỏ và ấu trùng của chúng thu được từ người và cá ở Việt Nam bao gồm mẫu HspMND2 (VN) có độ dài gen ITS-2 là 446 bp và mẫu HspMND (VN) có độ dài gen ITS- 2 là 290 bp. 3.3. Sự tương đồng của nucleotide trong gen ITS-2 Mức độ tương đồng các nucleotide được thể hiện trong Bảng 2. VIEÄN NGHIEÂN CÖÙU NUOÂI TROÀNG THUÛY SAÛN 2 60 TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 6 - THAÙNG 8/2015 Hình 3. So sánh trình tự nucleotide gen ITS-2 của các mẫu sán lá ruột nhỏ Haplorchis Việt Nam, Thái Lan và sán lá gan nhỏ (Clonorchis sinensis; Opisthorchis viverrini). Ghi chú: Sai khác trình tự của các chủng so với Hpu (TL1) biểu thị bằng chính ký hiệu nucleotide của chúng; dấu (.) biểu thị sự giống nhau; dấu (-) không có nucleotide tương ứng. VIEÄN NGHIEÂN CÖÙU NUOÂI TROÀNG THUÛY SAÛN 2 TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 6 - THAÙNG 8/2015 61 (VN) có độ dài gen ITS-2 là 446 bp, tương đương ITS-2 của H. taichui; và mẫu HspMND (VN) có độ dài gen ITS-2 là 290 bp, tương đương gen ITS-2 của H. pumilio. So sánh giữa các mẫu sán và ấu trùng thu được ở Việt Nam với H. taichui và H. pumilio của Thái Lan và so sánh với một số sán lá truyền lây khác như sán lá gan nhỏ (C. sinensis và O. viverrini) về mức độ tương đồng các nucleotide cho thấy mẫu H. pumilio ((HpuM (TL) nguồn gốc từ Thái Lan) phân tích ở nghiên cứu này và mẫu của Ando và ctv., (2001) (từ Thái Lan) có sự tương đồng nucleotide của gen ITS-2 là 96% và giữa mẫu HspMND (VN) với mẫu H. pumilio của Bảng 2. Sự tương đồng nucleotide trong gen ITS-2 ở một số loài sán lá truyền lây qua cá Ghi chú: 1: Hpu (TL1); 2: HpuM (TL); 3: Hpu (AY245706); 4: HspMND (VN); 5: HspND (VN); 6: HspNDV (VN); 7: Hta (TL1); 8: Hta (TL); 9: HspMND2 (VN); 10: Hta (AY245705); 11: Ovi (AY584735); 12: Ovi (TL1); 13: Csi (SKR) IV. THẢO LUẬN Từ kết quả giải trình tự gen cho thấy có sự sai khác về độ dài gen ITS-2 giữa H. taichui và H. pumilio. Đối với H. taichui, gen ITS- 2 có độ dài là 445-446 bp còn đối với mẫu sán H. pumilio là 290 bp. Gen ITS-2 của H. taichui dài hơn H. pumilio do có một đoạn 154 nucleotide từ nucleotide thứ 198 đến nucleotide 352 được chèn vào. Sự sai khác nhiều về trình tự sắp xếp các nucleotide giữa H. taichui và H. pumilio chủ yếu xảy ra ở các vị trí sau đoạn chèn, còn các vị trí trước đoạn chèn có sự tương đồng lớn. Các mẫu sán ruột nhỏ và ấu trùng của chúng thu được từ người và cá ở Việt Nam bao gồm mẫu HspMND2 VIEÄN NGHIEÂN CÖÙU NUOÂI TROÀNG THUÛY SAÛN 2 62 TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 6 - THAÙNG 8/2015 V. KẾT LUẬN Kết quả giải trình tự gen ITS2 sán lá ruột nhỏ Haplorchis spp. ở các giai đoạn cercariae, metacercariae và sán trưởng thành được thu từ ốc, cá, chó mèo, người ở Nam Định, Nghệ An Việt Nam và vùng Đông Bắc Thái Lan cho thấy có sự sai khác về độ dài đoạn gen ITS2 giữa sán lá ruột nhỏ loài H. taichui và H. pumilio. Đối với H. taichui, gen ITS2 có độ dài là 445 - 446 bp còn đối với mẫu sán H. pumilio là 290 bp. Giữa 2 loài sán lá ruột nhỏ H. taichui và H. pumilio có sự sai khác về độ dài và trật tự gen ITS-2 được thể hiện ở sự chèn đoạn gen 154 nucleotide, làm cho đoạn gen ITS-2 của H. taichui dài hơn đoạn gen ITS-2 của H. pumilio. Có rất ít sự sai khác về trình tự nucleotide trong gen ITS-2 của H. pumilio giữa mẫu sán của Việt Nam và mẫu sán của Thái Lan trong nghiên cứu này (tương đồng đạt 99%) và có sự sai khác không đáng kể giữa mẫu sán của Thái Lan trong nghiên cứu này với mẫu sán Thái Lan trong nghiên cứu của các tác giả khác. Có sự giống nhau hoàn toàn về trình tự sắp xếp nucleotide trong gen ITS-2 của H. taichui giữa mẫu sán Việt Nam và mẫu sán Thái Lan trong nghiên cứu này và giống nhau gần như tuyệt đối với mẫu sán Thái Lan của tác giả Ando và ctv., (2001). Nghiên cứu này giúp rút ngắn trong nghiên cứu vòng đời của sán lá ruột nhỏ nói riêng và sán lá song chủ nói chung. TÀI LIỆU THAM KHẢO Tài liệu tiếng Việt Kim Văn Vạn, Lê Thanh Hòa, Nguyễn Thị Bích Nga, Nguyễn Thị Tuyết Nhung và Anders Dalgaard, 2007a. Phân biệt sán lá ruột nhỏ Haplorchis taichui và H. pumilio với các loài sán lá khác sử dụng chỉ thị ITS-2 (internal transcribed spacer). Tạp chí Khoa học kỹ thuật nông nghiệp, Trường Đại học Nông nghiệp 1. Tập V số 1/2007. NXB Nông nghiệp, Trang 36-43. ISSN: 1895-0004. Thái Lan trong nghiên cứu này HpuM (TL) và mẫu phân tích của Ando và ctv., (2001) có sự tương đồng nucleotide tương ứng là 99 và 96%. Trong khi đó sự tương đồng nucleotide trong các mẫu H. pumilio của Thái Lan và cả mẫu HspMND (VN) so với Hpu (AY245706) từ Ngân hàng gen chỉ đạt 94%. Mức độ tương đồng nucleotide giữa các mẫu (1-4) đạt tỷ lệ tương đối cao (94-99%) tỷ lệ này phản ánh tương đồng thường thấy ở trong một loài. Như vậy có thể kết luận mẫu HspMND (VN) của Việt Nam là H. pumilio. Đối với mẫu H. taichui có nguồn gốc Thái Lan trong nghiên cứu này Hta (TL) với mẫu Hta (TL1) trong nghiên cứu của Ando và ctv., (2001) cho thấy có sự tương đồng nucleotide là 99%. Trong khi đó mẫu HspMND2 (VN) có sự tương đồng rất cao từ 99-100% so với mẫu H. taichui của Thái Lan và có sự tương đồng rất thấp với các mẫu H. pumilio chỉ đạt từ 52-54%, và đây là mức độ tương đồng thường thấy ở 2 loài khác nhau. Vì vậy có thể kết luận mẫu HspMND2 (VN) của Việt Nam là H. taichui. Đối với mẫu ký hiệu HspNDV (VN) khi phân tích sự tương đồng nucleotide vùng gen ITS-2 cho thấy tương đồng tuyệt đối 100% so với O. viverrini từ Ngân hàng gen. Le Thanh Hoa và ctv., (2003) cho rằng sán lá gan nhỏ O. viverrini chủ yếu phân bố ở vùng Nam Trung bộ và phía Nam, nhưng mẫu HspNDV (VN) chúng tôi phân tích ở đây lại được tìm thấy ở vùng Nam Định. Điều này cho thấy cần thiết có nghiên cứu bổ sung về nguồn gốc để có kết luận chắc chắn hơn. Trình tự gen ITS-2 của Hta (AY245705) thu thập từ Ngân hàng gen đem so sánh với mẫu nghiên cứu của chúng tôi và các mẫu tham khảo khác cho thấy có tỷ lệ tương đồng rất thấp (45%) với nhóm H. taichui và 54-68% với nhóm H. pumilio. Vậy chắc chắn trình tự này trong Ngân hàng gen là không đúng với H. taichui. VIEÄN NGHIEÂN CÖÙU NUOÂI TROÀNG THUÛY SAÛN 2 TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 6 - THAÙNG 8/2015 63 Kim Van Van, Anders Dalgaard, David Blair and Thanh Hoa Le, 2009. Haplorchis pumilio & H. taichui in Vietnam discriminated using ITS-2 DNA Se quence data from adults and larvae. Experimental Parasitology 123 Issue 2, October, 2009. Pages 141-151. ISSN 0014-4894 Kumar, S., Tamura, K., Nei, M., 2004. MEGA3: Integrated Software for Molecular Evolutionary Genetics Analysis and Sequence Alignment. Briefings in Bioinformatics 5, 150-163. Le, T.H., N.V., Chuong, N.V., De, P. Sithitharworn, N.B., Nga, T.N., Trung, L.K., Thuan, 2003. Report on molecular analysis of Opisthorchis viverrini collected from Phu Yen province. Proceedings of National Conference on Molecular Biology and Biochemistry, Hanoi (22-24.10.2003). Le, T.H., Blair, D., and McManus, D.P., 2002. Mitochondrial genomes of parasitic flatworms. Trends Parasitol, 18: 206-213. Nicholas, K.B., and H.B., Nicholas, 1999. GeneDoc: a tool for editting and annotating multiple sequence alignments. Distributed by authors. Tesana, S., Srisawangwonk, T., Kaekes, S., Sithithaworn, P., Kanla, P., Arunyanart, C., 1991. Egg shell morphology of the small eggs of human trematodes in Thailand. Southeast Asian J Trop Med Public Health 1991; 22: 631-6. LỜI CẢM ƠN Tác giả gửi lời cảm ơn Tổ chức DANIDA và dự án FIBOZOPA đã cung cấp kinh phí và tạo mọi điều kiện thuận lợi cho nghiên cứu này. Kim Văn Vạn, Lê Thanh Hòa, Nguyễn Văn Đề, Nguyễn Thị Bích Nga, Nguyễn Thị Tuyết Nhung và Anders Dalgaard, 2007b. Giám định sán lá ruột nhỏ Haplorchis taichui và H. pumilio sử dụng chỉ thị ITS-2 (internal transcribed spacer). Y học thành phố Hồ Chí Minh. Chuyên đề ký sinh trùng, Trường Đại học Y Dược TP. Hồ Chí Minh. Tập 11 *Phụ bản của số 2* 2007. Trang 104-110. Kim Văn Vạn, Lê Thanh Hòa, Nguyễn Văn Đề và Anders Dalgaard, 2007c. Ứng dụng sinh học phân tử xác định các giai đoạn phát triển của sán lá truyền lây qua cá từ các ký chủ trong vòng đời. Hội nghị chuyên đề chào mừng 105 năm thành lập trường ĐH Y Hà Nội: “Y-sinh học phân tử ứng dụng trong ngành ký sinh trùng”. Trường Đại học Y Hà Nội. Trang 84-91. Tài liệu tiếng Anh Ando, K., Sathithaworn, P., Nuchjungreed, C., Tesana, S., Srisawangwong, T., Limviroj, W., and Chinzei, Y., 2001. Nucleotide sequence of mitochondrial CO I and ribosomal ITS-2 genes of Opisthochis viverrini in Northeast Thailand. Southeast Asian J Trop Med Public Health 2001; 32: 17-22. Bowles, J., Blair, D., McManus, D.P., 1995. A molecular phylogeny of the human schistosomes. Mol Phylogenet Evol 4: 103- 109. Dzikowski, R., Levy, M.G., Poore, M. F., Flowers, J. R., and Paperna, I., 2004. Use of rDNA polymorphism for identification of Heterophyidae infecting freshwater fishes. Dis Aquat Org 2004; 59: 35-41. Kim Van Van and Dinh Thi Thuy, 2008. Comparison of Diagnostic Methods for the Detection of Parasites in Fish. Journal of Science and Development. Hanoi University of Agriculture. Special Issue April 2008. Agricultural Publishing House, Pages 136- 144. ISSN: 1895-0004. VIEÄN NGHIEÂN CÖÙU NUOÂI TROÀNG THUÛY SAÛN 2 64 TAÏP CHÍ NGHEÀ CAÙ SOÂNG CÖÛU LONG - 6 - THAÙNG 8/2015 APPLIED BIOLOGY TECHNOLOGY IN DIAGNOSIS PARASITE TRANSMIT VIA FISH Kim Van Van1* ABSTRACT Biotechnology was applied in general aquaculture and special diagnostic aquaculture pathogents. This paper will introduce applying biotechnology in zoonotic parasitological diagnostics as small intestine fluke: Haplorchis spp are tiny trematodes to be found in the small intestines of various definitive hosts such as human, birds, cats, dogs and rats. Human and other definitive hosts are infected by eating raw freshwater fishes containing encysted metacercariae. Haplorchis spp. is not easy to discriminate from other trematodes due to their minute size, similar morphology of eggs, cercaria, metacercaria and adult worm. A molecular method, for the first time in Vietnam, was applied to identify H. pumilio and H. taichui based on amplification of ITS-2 using forward primer, 3SF: 5’-GGTACCGGTGGATCACTCGGCTCGTG-3’ and reverse primer BD2R: 5’-TATGCTTAAATTCAGCGGGT-3-BD2R) with annealing at 50oC in the polymerase chain reaction (PCR) and sequencing for analyzing the nucleotide composition. Samples including adult worm and metacercariae were collected on human and fish in Nam Dinh (Vietnam) and Bangkok (Thailand). Length of ITS- 2 specific for H. taichui is 446bp; and for H. pumilio is 290bp. ITS-2 sequence in H. taichui longer than that in H. pumilio is due to the insertion of 154 nucleotides between nucleotide 198 and 352. There was high identity rate of nucleotides (99- 100%) in the ITS-2 gene between Vietnamese and Thai H. taichui or H. pumilii respectively. Keywords: Haplorchis spp, H. taichui, H. pumilio, PCR, identity. Người phản biện: TS. Đinh Thị Thủy Ngày nhận bài: 29/5/2015 Ngày thông qua phản biện: 03/8/2015 Ngày duyệt đăng: 07/8/2015 1Vietnam National University of Agriculture *Email: kvvan@vnua.edu.vn
File đính kèm:
 ung_dung_cong_nghe_sinh_hoc_trong_chan_doan_ky_sinh_trung_tr.pdf
ung_dung_cong_nghe_sinh_hoc_trong_chan_doan_ky_sinh_trung_tr.pdf

