Mô hình học sâu cho bài toán gán nhãn ngữ nghĩa trên văn bản y sinh
Chúng tôi xây dựng một mô hình gán nhãn Cấu trúc Đối số Vị ngữ cho văn bản Y Sinh. Cấu trúc
Đối số Vị ngữ là thông tin ngữ nghĩa quan trọng của văn bản, do nó chuyển tải sự kiện chính được
nói đến trong mỗi câu. Rút trích được Cấu trúc Đối số Vị ngữ trong câu là tiền đề quan trọng để
máy tính có thể giải quyết được hàng loạt bài toán khác liên quan đến ngữ nghĩa của văn bản như
rút trích sự kiện, rút trích thực thể, hệ hỏi đáp Cấu trúc Đối số Vị ngữ phụ thuộc vào lĩnh vực của
văn bản. Do đó, trong lĩnh vực Y Sinh, văn bản cần xác định khung Đối số Vị ngữ hoàn toàn mới so
với lĩnh vực tổng quát. Với đặc thù phải xử lý trên một khung đối số mới, việc xác định bộ đặc trưng
cho học máy là khó và đòi hỏi nhiều công sức chuyên gia. Để giải quyết thách thức này, chúng tôi
chọn huấn luyện mô hình của mình bằng phương pháp Học sâu (Deep learning) với Mạng nơ ron
bộ nhớ ngắn dài hai chiều (Bi-directional Long Short Term Memory). Học sâu là phương pháp học
máy không đòi hỏi con người phải xác định bộ đặc trưng một cách thủ công. Ngoài ra, chúng tôi
cũng tích hợp kết nối cao tốc (Highway Connection) giữa những tầng nơ ron ẩn không liên tiếp
để hạn chế mất mát đạo hàm. Bên cạnh đó, để khắc phục vấn đề ngữ liệu huấn luyện ít, chúng
tôi tích hợp Học sâu với kỹ thuật Học đa tác vụ. Học Đa tác vụ giúp cho tác vụ chính (bài toán gán
nhãn Cấu trúc Đối số Vị ngữ) được bổ trợ tri thức từ một tác vụ phụ có liên quan mật thiết là bài
toán rút trích Thực thể. Mô hình của chúng tôi đạt F1 = 72% mà không cần chuyên gia thiết kế bất
kỳ đặc trưng nào, qua đó cho thấy triển vọng của Học sâu trong bài toán này. Ngoài ra, kết quả
thực nghiệm cũng cho thấy Học đa tác vụ là kỹ thuật phù hợp để khắc phục vấn đề ngữ liệu huấn
luyện ít trong lĩnh vực Y Sinh vì nó cải thiện được độ đo F1.

Trang 1

Trang 2

Trang 3

Trang 4
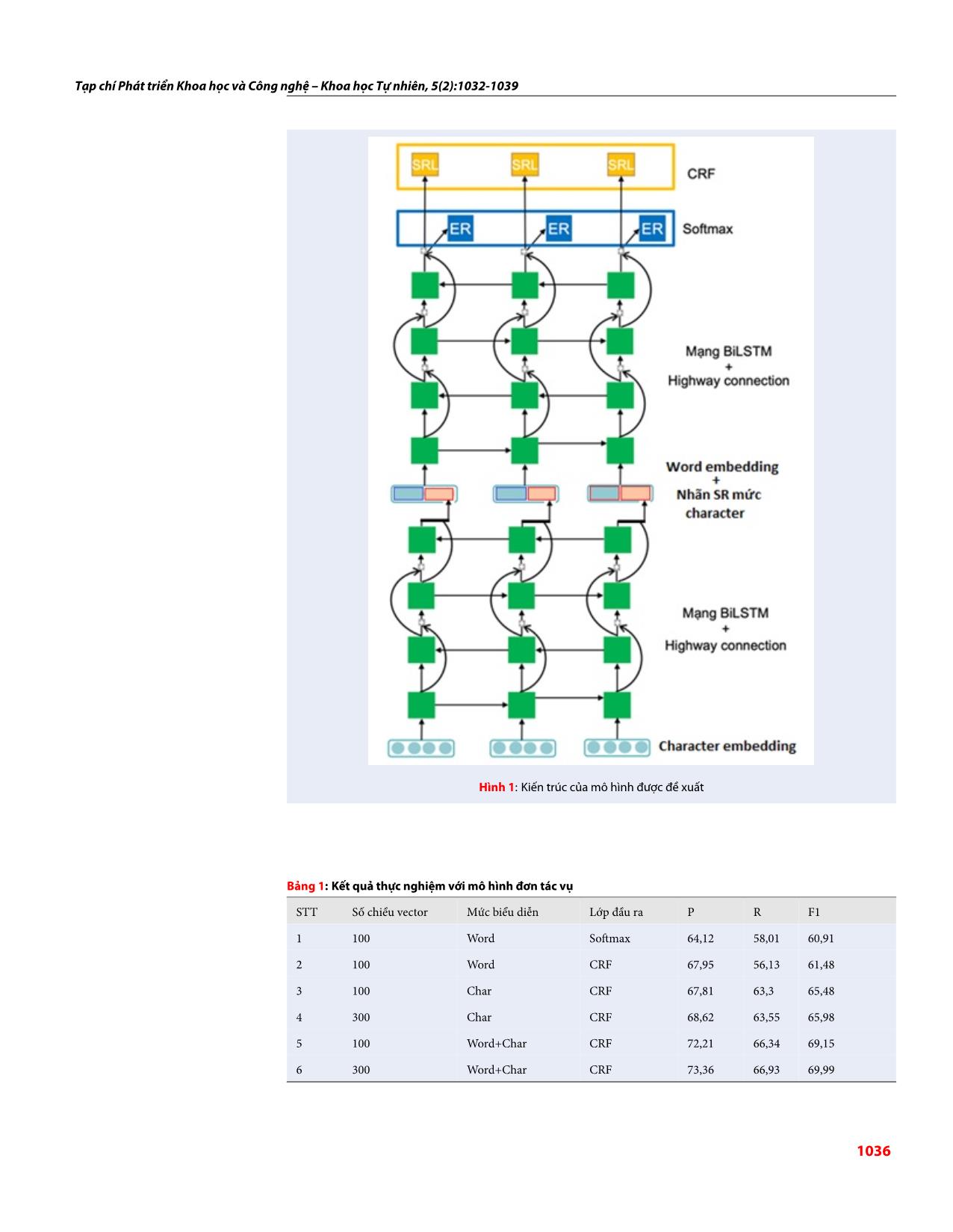
Trang 5

Trang 6

Trang 7

Trang 8
Tóm tắt nội dung tài liệu: Mô hình học sâu cho bài toán gán nhãn ngữ nghĩa trên văn bản y sinh

Khi áp cho thấy ở tầng đầu ra của tác vụ SRL, CRF là dụng vào văn bản Y Sinh, chúng tôi nhận thấy bài toán phù hợp hơn so với Softmax. SRL có liên quan mật thiết với bài toán Rút trích Thực thể (Named Entity Recognition – NER), vì loại thực KẾT LUẬN thể của đối số quyết định vai trò ngữ nghĩa của đối số (Ví dụ loại thực thể DNA chỉ có thể giữ vai trò “tác Chúng tôi đã xây đựng được mô hình học sâu cho bài nhân” của vị ngữ “encode” chứ không thể giữ vai trò toán SRL trên văn bản Y Sinh với một dữ liệu huấn “sản phẩm”). Vì vậy, chúng tôi chọn bài toán NER là luyện có kích thước hạn chế. Mô hình của chúng tôi tác vụ phụ trong mô hình học đa tác vụ của mình để trong lĩnh vực Y Sinh đạt F1 = 72% với chỉ 1389 câu bổ trợ cho tác vụ chính là SRL. trong dữ liệu huấn luyện. Kết quả này không cách quá Ngoài ra, DL kết hợp với học máy truyền thống sẽ xa so với kết quả cao nhất trong lĩnh vực tổng quát đạt cho kết quả tốt hơn từng kỹ thuật riêng lẻ 39. Do đó, F1 = 77% trong công trình tiên phong đề xuất học đa trong mô hình của chúng tôi, tầng đầu ra của mạng tác vụ cho bài toán SRL với dữ liệu huấn luyện hơn 36 nơ ron được phân loại một lần nữa bởi mô hình học 6000 câu . Kết quả thử nghiệm của chúng tôi cho máy truyền thống là CRF và Softmax (Hình 1). Kết thấy mô hình học đa tác vụ cũng phù hợp với SRL quả thực nghiệm của mô hình sẽ được phân tích trong trong lĩnh vực Y Sinh, một lĩnh vực còn hạn chế về mục Kết quả thực nghiệm. kích thước ngữ liệu gán nhãn sẵn. Hướng phát triển của chúng tôi là thử nghiệm kết hợp KẾT QUẢ THỬ NGHIỆM VÀ THẢO tri thức ngữ pháp với mức biểu diễn từ và ký tự để LUẬN làm giàu đặc trưng cho mô hình. Đồng thời, chúng Bộ ngữ liệu được sử dụng để huấn luyện và đánh giá tôi sẽ kết hợp kỹ thuật học chủ động (Active learn- là GREC, được xây dựng bởi trung tâm Text Mining ing) với học đa tác vụ để nâng cao hiệu quả của mô (NaCTeM),Khoa Khoa học Máy tính, Trường Đại học hình. Ngoài ra, chúng tôi sẽ nghiên cứu ứng dụng Manchester, Anh quốc 8. Ưu điểm của GREC là các vị transfer learning từ một mô hình pre-trained để hỗ ngữ của câu không chỉ có động từ mà còn bao gồm trợ mô hình học sâu khi tập dữ liệu huấn luyện có cả danh động nên độ phủ cao, với 4770 vị ngữ. Đồng kích thước hạn chế. thời GREC còn gán nhãn thực thể nên rất thuận lợi LỜI CẢM ƠN cho học đa tác vụ. Về phương pháp đánh giá, chúng tôi dùng phương pháp đánh giá chéo 10 pha (10-fold Nghiên cứu được tài trợ bởi Trường Đại học Khoa học cross validation) Tự nhiên, ĐHQG-HCM trong khuôn khổ Đề tài mã Chúng tôi thử nghiệm và so sánh kết quả của mô hình số CNTT 2020-13 với ba mức biểu diễn là chỉ có mức từ (word embed- DANH MỤC TỪ VIẾT TẮT ding), chỉ có mức ký tự (character embedding) và mức từ kết hợp với mức ký tự với những số chiều vector PAS: Cấu trúc Đối số Vị ngữ (Predicate Argument khác nhau. Chúng tôi cũng so sánh hiệu quả của Soft- Structure) max và CRF ở tầng đầu ra, cũng như so sánh hiệu quả SRL: Gán nhãn Ngữ nghĩa (Semantic Role Labelling) của mô hình khi không có học đa tác vụ (Bảng 1) và NER: Gán nhãn thực thể (Named Entity Recognition) khi có học đa tác vụ (Bảng 2). DL : Ứng dụng học sâu (Deep Learning) Từ kết quả thử nghiệm cho thấy: XUNG ĐỘT LỢI ÍCH TÁC GIẢ • F1 cao nhất của mô hình có học đa tác vụ cao Các tác giả tuyên bố rằng họ không có xung đột lợi hơn F1 cao nhất của mô hình học đơn tác vụ ích. 5.14%, đây là một khoảng cách đáng kể. Điều này củng cố giả thiết của chúng tôi về hiệu quả ĐÓNG GÓP CỦA CÁC TÁC GIẢ tích cực của học đa tác vụ cũng như việc lựa Tuấn Nguyên Hoài Đức chủ trì đề tài, tiến hành khảo chọn tác vụ phụ là NER đối với SRL cho văn bản sát hiện trạng, thu thập dữ liệu, phân tích đánh giá Y Sinh. giải pháp và viết bài. • Ở cả mô hình học đơn tác vụ và đa tác vụ đều Lê Đình Việt Huy và Trần Tiền Lợi Long Tứ tham gia cho thấy việc tăng số chiều vector không nâng khảo sát hiện trạng, đề xuất giải pháp và lập trình thử cao F1 đáng kể bằng việc chia mịn mức biểu nghiệm. diễn, từ mức từ thành mức ký tự. 1035 Tạp chí Phát triển Khoa học và Công nghệ – Khoa học Tự nhiên, 5(2):1032-1039 Hình 1: Kiến trúc của mô hình được đề xuất Bảng 1: Kết quả thực nghiệm với mô hình đơn tác vụ STT Số chiều vector Mức biểu diễn Lớp đầu ra P R F1 1 100 Word Softmax 64,12 58,01 60,91 2 100 Word CRF 67,95 56,13 61,48 3 100 Char CRF 67,81 63,3 65,48 4 300 Char CRF 68,62 63,55 65,98 5 100 Word+Char CRF 72,21 66,34 69,15 6 300 Word+Char CRF 73,36 66,93 69,99 1036 Tạp chí Phát triển Khoa học và Công nghệ – Khoa học Tự nhiên, 5(2):1032-1039 Bảng 2: Kết quả thực nghiệm với mô hình đa tác vụ. STT Số chiều vector Mức biểu Lớp đầu ra cho SRL Lớp đầu ra cho Kết quả SRL diễn NER R F1 1 100 Word Softmax CRF 68,93 64,31 66,54 2 100 Word CRF CRF 69,27 64,97 67,05 3 100 Word CRF Softmax 70,04 67,74 68,87 4 100 Char CRF Softmax 73,29 67,97 70,53 5 300 Char CRF Softmax 74,57 67,90 72,08 6 100 Word+Char CRF Softmax 78,03 70,97 74,33 7 300 Word+Char CRF Softmax 78,86 71,74 75,13 TÀI LIỆU THAM KHẢO 17. Grenager T, et al. Manning, Unsupervised Discovery of a Sta- tistical Verb Lexicon. EMNLP. 2007;06:1–8. Available from: 1. Enderle JD, et al. Introduction to Biomedical Engineering, Aca- https://doi.org/10.3115/1610075.1610077. demic Press. 2012;p. 16–21. 18. Wattarujeekrit T. Exploring Predicate-Argument Relations for 2. Schmidhuber J. Deep Learning in Neural Networks: Named Entity Recognition in the Molecular Biology Domain, An Overview, Neural Networks. 2015;61:85–117. PMID: International Conference on Discovery Science. 2005;8:267– 25462637. Available from: https://doi.org/10.1016/j.neunet. 280. Available from: https://doi.org/10.1007/11563983_23. 2014.09.003. 19. Stevens G. XARA: An XML- and rule-based semantic role 3. Johnson CR, et al. The FrameNet project: Tools for lexi- labeler, The Linguistic Annotation Workshop, Annual Meet- con building, International Conference on Computational Lin- ing of the Association for Computational Linguistics. 2007;45. guistics. 1998;17:86–90. PMID: https://doi.org/10.3115/1642059.1642077. Available 4. Kipper K, et al. Class-based construction of a verb lexicon, from: 26110305. AAAI-2000. 2000;(2000):691–696. 20. Iida R, et al. Annotating a Japanese Text Corpus with 5. Kingsbury P, Palmer M. From Treebank to PropBank, Inter- Predicate-Argument and Coreference Relations, the Linguis- national Conference on Language Resources and Evaluation. tic Annotation Workshop. 2007;p. 132–139. Available from: 2002;12:38–43. https://doi.org/10.3115/1642059.1642081. 6. Chou WC, et al. A semi-automatic method for annotating a 21. Riloff E. Automatically Generating Extraction Patterns from biomedical proposition bank, The workshop on frontiers in Untagged Text, National Conference on Artificial Intelligence. linguistically annotated corpora. 2006;p. 5–12. 1996;19:1044–1049. 7. Wattarujeekrit T, et al. PASBio: predicate-argument structures 22. Riloff E. An empirical approach to conceptual case frame ac- for event extraction in molecular biology, BMC Bioinformatics. quisition, The Workshop on Very Large Corpora. 1998;6:49–56. 2004;5:155–163. PMID: 15494078. Available from: https://doi. 23. Riloff E. Automatically constructing a dictionary for informa- org/10.1186/1471-2105-5-155. tion extraction tasks, National Conference on Artificial Intelli- 8. Thompson P, Cotter P, McNaught J, et al. Building a Bio-Event gence (AAAI). 1993;1:811–816. Annotated Corpus for the Acquisition of Semantic Frames 24. Huang M. Discovering patterns to extract protein-protein in- from Biomedical Corpora. LREC. 2008;. teractions from full texts. Bioinformatics. 2004;p. 3604–3612. 9. Pollard C, Sag IA. Head-Driven Phrase Structure Grammar. IL: PMID: 15284092. Available from: https://doi.org/10.1093/ Univ. of Chicago Press. 1994;. bioinformatics/bth451. 10. Liakata M, et al. From Trees To Predicate-Argument Struc- 25. Blaheta D, Charniak E. Assigning function tags to parsed text, tures, International Conference on Computational Linguis- the Annual Meeting of the North American Chapter of the tics. 2002;20:563–569. Available from: https://doi.org/10.3115/ ACL. 2000;1:234–240. 1072228.1072333. 26. Gildea D, Jurafsky D. Automatic labeling of semantic roles, 11. Marcus M, et al. The Penn Treebank: Annotating Predicate Ar- Computational Linguistics. 2002;p. 245–288. Available from: gument Structure, The Human Language Technology Work- https://doi.org/10.1162/089120102760275983. shop. Plainsboro, NJ, 114119. 1994;Available from: https://doi. 27. Gildea D, Palmer M. The necessity of parsing for predicate ar- org/10.3115/1075812.1075835. gument recognition, Meeting of the Association for Compu- 12. Carreras X, Màrquez L. Introduction To the CoNLL-2005 shared tational Linguistics. 2002;40:239–246. Available from: https: task: Semantic role labeling, CoNLL. 2005;p. 152–164. Avail- //doi.org/10.3115/1073083.1073124. able from: https://doi.org/10.3115/1706543.1706571. 28. Surdeanu M, Harabagiu S, et al. Using Predicate-Argument 13. Carreras X, Màrquez L. Introduction to the CoNLL-2004 shared Structure for Information Extraction, Annual Conference on task: Semantic role labeling, HLT-NAACL 2004 Workshop 8th the Association for Computational Linguistics. 2013;41:46–51. Conf. Comput. Natural Lang. Learn. 2004;p. 89–97. 29. Kingsbury P, Palmer M, Marcus M. Adding Semantic Annota- 14. Park KM, et al. Two-phase semantic role labeling based on tion to the Penn TreeBank, The Human Language Technology support vector machines, CoNLL. 2004;. Conference. 2002;p. 252–256. 15. Surdeanu M, et al. Semantic role labeling using complete syn- 30. Tsai RTH, et al. BIOSMILE: Adapting Semantic Role Labeling for tactic analysis, CoNLL. 2005;p. 67–72. Available from: https: Biomedical Verbs: An Exponential Model Coupled with Auto- //doi.org/10.3115/1706543.1706586. matically Generated Template Features, BioNLP Conference. 16. Chi-San (Althon) Lin, Tony C. Smith, Semantic role labeling via 2006;. consensus in pattern-matching, CONLL. 2005;5:185–188. 31. Swier RS, Stevenson S. Exploiting a Verb Lexicon in Automatic Semantic Role Labelling, EMNLP. 2005;05:883–890. 1037 Tạp chí Phát triển Khoa học và Công nghệ – Khoa học Tự nhiên, 5(2):1032-1039 32. Swier RS, Stevenson S. Unsupervised Semantic Role Labeling, Resource NLP. 2018;p. 43–50. Available from: https://doi.org/ EMNLP. 2004;04:95–102. 10.18653/v1/W18-3406. 33. Bengio Y, Simard P. Problem of learning long-term Dependen- 37. Zhou J, Xu W. End-to-end Learning of Semantic Role La- cies in Recurrent Network, IEEE Transactions on Neural Net- beling Using Recurrent Neural Networks, Annual Meeting works archive. 1994;5:157–166. PMID: 18267787. Available of the Association for Computational Linguistics 53 - Inter- from: https://doi.org/10.1109/72.279181. national Joint Conference on Natural Language Processing. 34. Hochreiter S. Long-Short Term Memory, Neural Computation 2015;7:1127–1137. Available from: https://doi.org/10.3115/v1/ Archive. 1997;9:1735–1780. PMID: 9377276. Available from: P15-1109. https://doi.org/10.1162/neco.1997.9.8.1735. 38. He L, et al. Deep Semantic Role Labeling: What Works and 35. Graves A, rahman Mohamed A, Hinton G. Speech Recog- What’s Next, Annual Meeting of the Association for Compu- nition with Deep Recurrent Neural networks, 1988. ICASSP. tational Linguistics. 2017;55:473–483. Available from: https: 2013;88:90–95. Available from: https://doi.org/10.1109/ //doi.org/10.18653/v1/P17-1044. ICASSP.2013.6638947. 39. Bethard YV. A survey on recent advances in named entity 36. Ikhwantri F, et al. Multi-Task Active Learning for Neu- recognition from deep learning models, International Confer- ral Semantic Role Labeling on Low Resource Conversational ence on Computational Linguistics. 2018;27:2145 –2158. Corpus, Workshop on Deep Learning Approaches for Low- 1038 Tạp chí Phát triển Khoa học và Công nghệ – Natural Sciences, 5(2):1032-1039 Open Access Full Text Article Research article A deep-learning model for semantic role labelling in medical documents Tuan Nguyen Hoai Duc1,*, Le Dinh Viet Huy2, Tran Tien Loi Long Tu3 TÓM TẮT We built a model labelling the Predicate Argument Structure (PAS)for biomedical documents. PASis an important semantic information of any document, because it reveals the main event mentioned Use your smartphone to scan this in each sentence. Extracting PAS in a sentence is an important premise for the computer to solve QR code and download this article a series of other problems related to the semantics in text such as event extraction, named entity extraction, question answering system The predicate argument structure is domain dependent. Therefore, in Biomedical field, it is required to define a completely new Predicate Argument frame compared to the general field. For a machine learning model to work well with a new argument frame, identifying a new feature set is required. This is difficult, manual and requires a lot of expert labor. To address this challenge, we chose to train our model with Deep Learning method utilizing Bi-directional Long Short Term Memory. Deep learning is a machine learning method that does not require defining the feature sets manually. In addition, we also integrate Highway Connection between hidden neuron layers to minimize derivative loss. Besides, to overcome the problem of small training corpus, we integrate Deep Learning with Multi-task Learning technique. Multi-task Learning helps the main task (PAS tagging) to be complemented with knowledge learnt from a closely related task, the NER. Our model achieved F1 = 75.13% without any manually designed feature, thereby showing the prospect of Deep Learning in this domain. In addition, the experiment results also show that Multi-task Learning is an appropriate technique to overcome the problem of little training data in biomedical fields, by improving the F1 score. Từ khoá: predicate argument structure, semantic role labelling, deep learning 1Faculty of Information Technology, University of Sciences, VNU-HCM, Vietnam. 2ZAMO LLC Technology Ltd. Company, Vietnam. 3Gameloft Vietnam Company, Vietnam. Liên hệ Tuan Nguyen Hoai Duc, Faculty of Information Technology, University of Sciences, VNU-HCM, Vietnam. Email: tnhduc@fit.hcmus.edu.vn Lịch sử • Ngày nhận: 18-7-2020 • Ngày chấp nhận: 01-4-2021 • Ngày đăng: 16-4-2021 DOI : 10.32508/stdjns.v5i2.928 Bản quyền © ĐHQG Tp.HCM. Đây là bài báo công bố mở được phát hành theo các điều khoản của the Creative Commons Attribution 4.0 International license. Trích dẫn bài báo này: Duc T N H, Huy L D V, Tu T T L L. A deep-learning model for semantic role labelling in medical documents. Sci. Tech. Dev. J. - Nat. Sci.; 5(2):1032-1039. 1039
File đính kèm:
 mo_hinh_hoc_sau_cho_bai_toan_gan_nhan_ngu_nghia_tren_van_ban.pdf
mo_hinh_hoc_sau_cho_bai_toan_gan_nhan_ngu_nghia_tren_van_ban.pdf

