Thiết kế hệ thống cấu trúc vector CRISPR/Cas9 để chỉnh sửa gen GmNAC29 liên quan tới khả năng chống chịu hạn của cây đậu tương
Đậu tương (Glycine max L.) là một trong những cây họ đậu có giá trị kinh tế cao được trồng rộng rãi ở
nhiều nơi trên thế giới. Tuy nhiên, đậu tương lại là cây trồng nhạy cảm với stress hạn. Các yếu tố phiên mã
NAC được biết là có tham gia vào quá trình điều hòa đáp ứng và chống chịu của cây trồng với stress hạn.
GmNaC29 được biết tới như là một gen đóng vai trò tiêu cực trong quá trình đáp ứng bất lợi trong đó có
hạn. Do vậy, trong nghiên cứu này, đã tiến hành thiết kế hệ thống vector chỉnh sửa gen CRISPR/Cas9 có
chứa các sgRNA cho chỉnh sửa gen GmNAC29. Kết quả này sẽ cung cấp hệ thống gây đột biến có định
hướng gen GmNAC29 liên quan tới khả năng chống chịu hạn của cây đậu tương

Trang 1

Trang 2

Trang 3
Trang 4

Trang 5

Trang 6

Trang 7
Bạn đang xem tài liệu "Thiết kế hệ thống cấu trúc vector CRISPR/Cas9 để chỉnh sửa gen GmNAC29 liên quan tới khả năng chống chịu hạn của cây đậu tương", để tải tài liệu gốc về máy hãy click vào nút Download ở trên
Tóm tắt nội dung tài liệu: Thiết kế hệ thống cấu trúc vector CRISPR/Cas9 để chỉnh sửa gen GmNAC29 liên quan tới khả năng chống chịu hạn của cây đậu tương

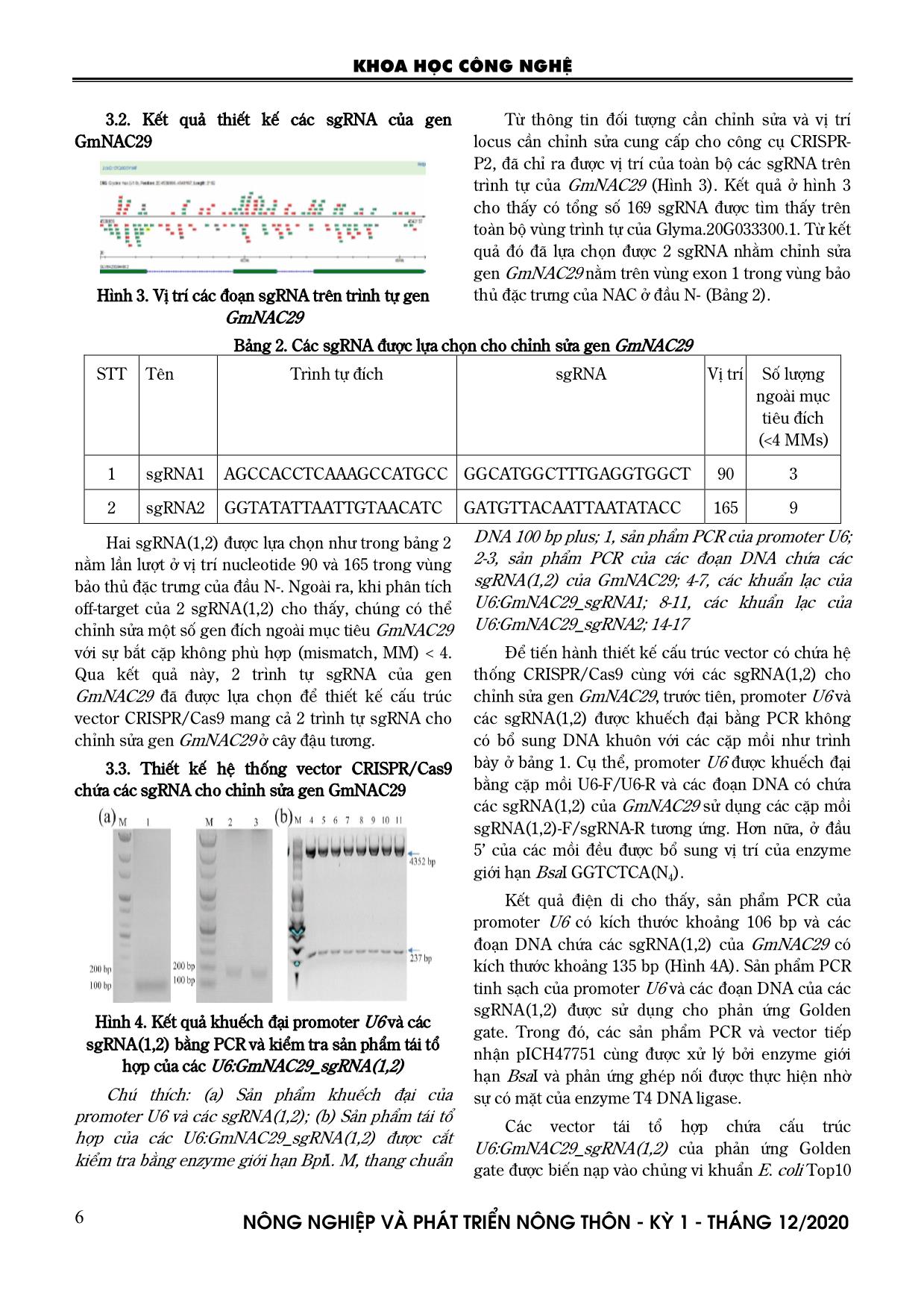
vùng exon và intron. Trong đó, Glyma.20G033300.1 có chứa 3 vùng exon có kích thước khác nhau để mã hóa liên tục cho gen GmNAC29 có chiều dài 843 bp (Hình 1). Hình 2. Kết quả phân tích trình tự DNA và protein của GmNAC29 Chú thích: (a) cấu trúc của GmNAC29; (b) Vùng bảo thủ của GmNAC29 ở đầu N- Tiếp theo, khi phân tích trình tự DNA của GmNAC29 trên web https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb .cgi, kết quả cho thấy GmNAC29 mã hóa cho 281 amino axit nằm ở phân họ NAM (No Apical Meristem) của họ yếu tố phiên mã NAC (Hình 2a). Hơn nữa, khi phân tích trình tự protein của GmNAC29 ở đầu N-, kết quả cho thấy GmNAC29 có chứa 5 vùng bảo thủ (A-E) và các vùng bảo thủ này tương đồng với một số protein NAC ở một số đối tượng cây trồng khác, cụ thể là cây cải xoong. Qua kết quả tìm kiếm này cho thấy, GmNAC29 là một gen thuộc họ yếu tố phiên mã NAC có chiều dài là 843 bp và mã hóa cho 281 amino axit. Từ đây, đã lựa chọn gen GmNAC29 là đối tượng cần chỉnh sửa và sử dụng trình tự của nó để thiết kế các sgRNA cho xây dựng hệ thống vector CRISPR/Cas9 mang sgRNA để chỉnh sửa gen GmNAC29. KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 1 - TH¸NG 12/2020 6 3.2. Kết quả thiết kế các sgRNA của gen GmNAC29 Hình 3. Vị trí các đoạn sgRNA trên trình tự gen GmNAC29 Từ thông tin đối tượng cần chỉnh sửa và vị trí locus cần chỉnh sửa cung cấp cho công cụ CRISPR- P2, đã chỉ ra được vị trí của toàn bộ các sgRNA trên trình tự của GmNAC29 (Hình 3). Kết quả ở hình 3 cho thấy có tổng số 169 sgRNA được tìm thấy trên toàn bộ vùng trình tự của Glyma.20G033300.1. Từ kết quả đó đã lựa chọn được 2 sgRNA nhằm chỉnh sửa gen GmNAC29 nằm trên vùng exon 1 trong vùng bảo thủ đặc trưng của NAC ở đầu N- (Bảng 2). Bảng 2. Các sgRNA được lựa chọn cho chỉnh sửa gen GmNAC29 STT Tên Trình tự đích sgRNA Vị trí Số lượng ngoài mục tiêu đích (<4 MMs) 1 sgRNA1 AGCCACCTCAAAGCCATGCC GGCATGGCTTTGAGGTGGCT 90 3 2 sgRNA2 GGTATATTAATTGTAACATC GATGTTACAATTAATATACC 165 9 Hai sgRNA(1,2) được lựa chọn như trong bảng 2 nằm lần lượt ở vị trí nucleotide 90 và 165 trong vùng bảo thủ đặc trưng của đầu N-. Ngoài ra, khi phân tích off-target của 2 sgRNA(1,2) cho thấy, chúng có thể chỉnh sửa một số gen đích ngoài mục tiêu GmNAC29 với sự bắt cặp không phù hợp (mismatch, MM) < 4. Qua kết quả này, 2 trình tự sgRNA của gen GmNAC29 đã được lựa chọn để thiết kế cấu trúc vector CRISPR/Cas9 mang cả 2 trình tự sgRNA cho chỉnh sửa gen GmNAC29 ở cây đậu tương. 3.3. Thiết kế hệ thống vector CRISPR/Cas9 chứa các sgRNA cho chỉnh sửa gen GmNAC29 Hình 4. Kết quả khuếch đại promoter U6 và các sgRNA(1,2) bằng PCR và kiểm tra sản phẩm tái tổ hợp của các U6:GmNAC29_sgRNA(1,2) Chú thích: (a) Sản phẩm khuếch đại của promoter U6 và các sgRNA(1,2); (b) Sản phẩm tái tổ hợp của các U6:GmNAC29_sgRNA(1,2) được cắt kiểm tra bằng enzyme giới hạn BpiI. M, thang chuẩn DNA 100 bp plus; 1, sản phẩm PCR của promoter U6; 2-3, sản phẩm PCR của các đoạn DNA chứa các sgRNA(1,2) của GmNAC29; 4-7, các khuẩn lạc của U6:GmNAC29_sgRNA1; 8-11, các khuẩn lạc của U6:GmNAC29_sgRNA2; 14-17 Để tiến hành thiết kế cấu trúc vector có chứa hệ thống CRISPR/Cas9 cùng với các sgRNA(1,2) cho chỉnh sửa gen GmNAC29, trước tiên, promoter U6 và các sgRNA(1,2) được khuếch đại bằng PCR không có bổ sung DNA khuôn với các cặp mồi như trình bày ở bảng 1. Cụ thể, promoter U6 được khuếch đại bằng cặp mồi U6-F/U6-R và các đoạn DNA có chứa các sgRNA(1,2) của GmNAC29 sử dụng các cặp mồi sgRNA(1,2)-F/sgRNA-R tương ứng. Hơn nữa, ở đầu 5’ của các mồi đều được bổ sung vị trí của enzyme giới hạn BsaI GGTCTCA(N4). Kết quả điện di cho thấy, sản phẩm PCR của promoter U6 có kích thước khoảng 106 bp và các đoạn DNA chứa các sgRNA(1,2) của GmNAC29 có kích thước khoảng 135 bp (Hình 4A). Sản phẩm PCR tinh sạch của promoter U6 và các đoạn DNA của các sgRNA(1,2) được sử dụng cho phản ứng Golden gate. Trong đó, các sản phẩm PCR và vector tiếp nhận pICH47751 cùng được xử lý bởi enzyme giới hạn BsaI và phản ứng ghép nối được thực hiện nhờ sự có mặt của enzyme T4 DNA ligase. Các vector tái tổ hợp chứa cấu trúc U6:GmNAC29_sgRNA(1,2) của phản ứng Golden gate được biến nạp vào chủng vi khuẩn E. coli Top10 KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 1 - TH¸NG 12/2020 7 bằng phương pháp sốc nhiệt. Dịch khuẩn sau khi biến nạp được cấy trải và nuôi qua đêm trên đĩa môi trường LB đặc có bổ sung 20 mg/L X-Gal kết hợp với 125 mg/L ampicillin ở 37oC. Bốn khuẩn lạc màu trắng cho mỗi cấu trúc U6:GmNAC29_sgRNA được lựa chọn để nuôi tăng sinh khối trong môi trường LB lỏng có bổ sung 125 mg/L Ampicilin để tách chiết thu DNA plasmid. Các vector tái tổ hợp cho mỗi U6:GmNAC29_sgRNA được cắt kiểm tra bằng enzyme giới hạn BpiI. Kết quả điện di cho thấy, với mỗi cấu trúc tái tổ hợp U6:GmNAC29_sgRNA sau khi cắt đều có 2 vạch băng trên bản gel. Cụ thể, 1 băng có kích thước khoảng 4352 bp trùng với kích thước của vector pICH47751 và 1 băng có kích thước 237 bp được dự đoán là của các cấu trúc U6:GmNAC29_sgRNA(1,2) (Hình 4B). Như vậy, bước đầu cho thấy việc tách dòng và ghép nối các đoạn sgRNA(1,2) của GmNAC29 và promoter U6 vào vector tiếp nhận pICH47751 đã thành công. Tiếp theo, 2 trong số 4 khuẩn lạc dương tính với mỗi cấu trúc U6:GmNAC29_sgRNA được lựa chọn để giải trình tự với mồi Lv11-sF1: 5’- GGTGTAAACAAATTGACGCTTAGA-3’ bằng máy giải trình tự Sanger. Bảng 3. Kết quả giải trình tự các cấu trúc U6:GmNAC29_sgRNA(1,2) U6:GmNAC29_sgRNA1 U6:GmNAC29_sgRNA2 tctgtGAAGACaaACTAgaattcccatggggagTgatcaaaag tcccacatcgatcaggtgatatatagcagcttagtttatataatgataga gtcgacatagcgattgGGCATGGCTTTGAGGTGGCTgtt ttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttg aaaaagtggcaccgagtcggtgctttttttctagacccagctttcttgtac aaagttggcattacgctTTACttGTCTTCtgcac TctgtGAAGACaaTTACgaattcccatggggagTgatcaaaa gtcccacatcgatcaggtgatatatagcagcttagtttatataatgatag agtcgacatagcgattgGATGTTACAATTAATATACCgt tttagagctagaaatagcaagttaaaataaggctagtccgttatcaactt gaaaaagtggcaccgagtcggtgctttttttctagacccagctttcttgt acaaagttggcattacgctCAGAttGTCTTCtgcac Chú thích: Chữ in đậm màu đỏ, vị trí enzyme giới hạn BsaI nhận biết; chữ in đậm màu xanh, vị trí cắt của enzyme giới hạn BsaI; chữ gạch chân, trình tự các sgRNA. Kết quả giải trình tự cho thấy, tất cả 2 vector pICH47751 có chứa các cấu trúc U6:GmNAC29_sgRNA(1,2) và đều mang trình tự chính xác của promoter U6 và theo sau là các sgRNA(1,2) của GmNAC29 (Bảng 3). Hình 5. Kết quả kiểm tra vector tái tổ hợp pID:U6:GmNAC29_sgRNA(1,2) bằng enzyme giới hạn BamHI Chú thích: M, 1Kb plus ladder; 1-3, DNA plasmid của các khuẩn lạc pID:U6:GmNAC29_sgRNA(1,2). Tiếp theo, cả 2 vector pICH47751 chứa các cấu trúc U6:GmNAC29_sgRNA(1,2) được sử dụng để ghép nối các cấu trúc này vào vector binary pID trong phản ứng Golden gate. Sản phẩm tái tổ hợp của cấu trúc vector pID:U6:GmNAC29_sgRNA(1,2) được biến nạp vào chủng vi khuẩn E. coli Top10. Sau đó, dịch khuẩn được cấy trải và nuôi qua đêm trên đĩa LB có bổ sung 50 mg/L kháng sinh kanamycin ở 37oC. Ba trong số các khuẩn lạc trắng được lựa chọn để tiếp tục nuôi tăng sinh khối trong LB lỏng có bổ sung 50 mg/L kanamycin cho tách chiết và tinh sạch DNA plasmid phục vụ cho việc giải trình tự và cắt kiểm tra bằng enzyme giới hạn. Kết quả cắt kiểm tra vector tái tổ hợp pID:U6:GmNAC29_sgRNA(1,2) bằng enzyme giới hạn BamHI cho thấy, các sản phẩm cắt đều có 2 băng; trong đó, 1 băng có kích thước khoảng 7738 bp có chứa vùng trình tự của 2 tổ hợp cấu trúc U6:GmNAC29_sgRNA(1,2), trùng với kích thước dự đoán và 1 băng có kích thước khoảng 4039 bp có chứa trình tự của gen Bar và Cas9 (Hình 5). Để đảm bảo sự chính xác, hai trong số ba khuẩn lạc đã được lựa chọn để giải trình tự kiểm tra lại. Kết quả cho thấy, vector pID chứa 2 tổ hợp cấu trúc U6:GmNAC29_sgRNA(1,2) có trình tự hoàn toàn KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 1 - TH¸NG 12/2020 8 chính xác như trình bày ở bảng 3 và được sắp xếp như trong hình 6. Hình 6. Cấu trúc vector pID:U6:GmNAC29_sgRNA(1,2) và tổ hợp cấu trúc các U6:GmNAC29_sgRNA(1,2) cho chỉnh sửa gen GmNAC29 Chú thích: (a) Cấu trúc vector pID:U6:GmNAC29_sgRNA(1,2). (b) Tổ hợp cấu trúc chứa các sgRNA của GmNAC29. LB, biên trái; NOSp- BAR-NOST, chỉ thị gen bar kháng glufosinate; 2x35S- hSpCas9-tNOS, Cas9 được điều khiển bởi promoter 35S; U6:GmNAC29_sgRNA(1,2), vị trí lần lượt của các sgRNA của GmNAC29 từ trái sang phải; RB, biên phải; pVS1, vùng bắt đầu sao chép; nptII, chỉ thị gen nptII kháng kanamycin. Qua hình 6 cho thấy, vector binary pID có chứa tổ hợp cấu trúc cho chỉnh sửa gen GmNAC29 ở cây đậu tương được sắp xếp lần lượt từ vùng LB sang RB gồm có: 1 vùng chứa đoạn gen Bar, 1 vùng chứa Cas9 được điều khiển bằng promoter 2x35S, và 2 sgRNA được điều khiển bởi promoter U6 riêng biệt, cùng với hệ thống chọn lọc đối với vi khuẩn là kháng sinh kanamycin và thực vật là glufosinate. Với kết quả này, cấu trúc vector binary pID có chứa hệ thống CRISPR/Cas9 và các U6:GmNAC29_sgRNA(1,2) cho chỉnh sửa gen GmNAC29 ở cây đậu tương đã được thiết kế thành công. Kỹ thuật chỉnh sửa gen bằng công nghệ CRISPR/Cas9 được biết tới là một công cụ hữu ích trong nghiên cứu chức năng cụ thể của một gen, gần đây hơn kỹ thuật này cũng đã được ứng dụng trong nghiên cứu chỉnh sửa các gen liên quan tới các đặc tính chống chịu với các điều kiện môi trường bất lợi khác nhau trên một số đối tượng cây trồng quan trọng (Bao et al., 2019). 4. KẾT LUẬN Các kết quả của nghiên cứu chỉ ra rằng, hệ thống vector CRISPR/Cas9 chứa tổ hợp các U6:sgRNA(1,2) cho chỉnh sửa có định hướng gen GmNAC29 đã được thiết kế thành công. Thành công của nghiên cứu này sẽ cung cấp hệ thống vector CRISPR/Cas9 cho chỉnh sửa gen đích GmNAC29 ở cây đậu tương nhằm tăng cường khả năng chống chịu hạn. LỜI CẢM ƠN Công trình nghiên cứu được thực hiện trong khuôn khổ đề tài “Nghiên cứu bước đầu tạo cây đậu tương tăng cường khả năng chịu hạn bằng kỹ thuật CRISPR/Cas9” theo Quyết định số 617/QĐ-KHNN- KH ngày 31/7/2019 của Giám đốc Viện Khoa học Nông nghiệp Việt Nam về việc Phê duyệt danh mục các nhiệm vụ thường xuyên Phòng Thí nghiệm Trọng điểm Công nghệ tế bào thực vật thực hiện từ năm 2019. Công trình nghiên cứu được đồng tài trợ bởi Quỹ Nghiên cứu Quốc gia Hàn Quốc (Mã số Đề tài NRF 2020M3A9I4038352 và 2020R1A6A1A03044344). TÀI LIỆU THAM KHẢO 1. Bao A., Burritt D. J., Chen H., Zhou X., Cao D., & Tran, L. P. 2019. The CRISPR/Cas9 system and its applications in crop genome editing. Crit Rev Biotechnol, 39(3): 321-336. 2. Basu S., Ramegowda V., Kumar A., Pereira A. (2016). Plant adaptation to drought stress. F1000Res, 5: 1554. 3. Nguyễn Hữu Kiên, Nguyễn Văn Đồng (2016). Yếu tố phiên mã NAC trong thực vật: vai trò và tiềm năng ứng dụng. Hội thảo Quốc gia về Khoa học cây trồng lần thứ hai, 2: 302-306. 4. Nuruzzaman M., Sharoni A. M., Kikuchi S. (2013). Roles of NAC transcription factors in the regulation of biotic and abiotic stress responses in plants. Front. Microbiol, 4: 248. 5. Puranik S., Sahu P. P., Srivastava P. S., Prasad M. (2012). NAC proteins: regulation and role in stress tolerance. Trends Plant Sci, 17: 369–381. 6. Ohbayashi I., Sugiyama M. (2018). Plant nucleolar stress response, a new face in the NAC- dependent cellular stress responses. Front. Plant Sci, 8: 2247. 7. Singh D., Yadav N. S., Tiwari V., Agarwal P. K., Jha B. (2016). A SNARE-like superfamily protein KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 1 - TH¸NG 12/2020 9 SbSLSP from the halophyte salicornia brachiata confers salt and drought tolerance by maintaining membrane stability, K(+)/Na(+) ratio, and antioxidant machinery. Front. Plant Sci, 7: 737. 8. Song L., Prince S., Valliyodan B., Joshi T., Maldonado dos Santos J. V., Wang J., Lin L., Wan J., Wang Y., Xu D., Nguyen H. T. (2016). Genome-wide transcriptome analysis of soybean primary root under varying water-deficit conditions. BMC Genomics, 17: 57. 9. Tak H., Negi S., Ganapathi T. R. (2017). Banana NAC transcription factor MusaNAC042 is positively associated with drought and salinity tolerance. Protoplasma, 254: 803–816. 10. Thirumalaikumar V. P., Devkar V., Mehterov N., Ali S., Ozgur R., Turkan I., Mueller-Roeber B., Balazadeh S. (2017). NAC transcription factor JUNGBRUNNEN1 enhances drought tolerance in tomato. Plant Biotechnol. J, 16: 354–366. 11. Valliyodan B., Ye H., Song L., Murphy M., Shannon J. G., Nguyen H. T. (2017). Genetic diversity and genomic strategies for improving drought and waterlogging tolerance in soybeans. J. Exp. Bot, 68: 1835–1849. 12. Wang F., Chen H. W., Li Q. T., Wei W., Li W., Zhang W. K., Ma B., Bi Y. D., Lai Y. C., Liu X. L., Man W. Q., Zhang J. S., Chen S. Y. (2015). GmWRKY27 interacts with GmMYB174 to reduce expression of GmNAC29 for stress tolerance in soybean plants. The Plant Journal, 83(2): 224-236. 13. Werner S., Engler C., Weber E., Gruetzner R., Marillonnet S. (2012). Fast track assembly of multigene constructs using Golden Gate cloning and the MoClo system. Bioeng Bugs, 3(1): 38-43. CONSTRUCTION OF THE CRISPR/Cas9 VECTOR SYSTEM FOR EDITING GmNAC29 GENE RELATED TO DROUGHT TOLERANCE IN SOYBEAN Nguyen Huu Kien1,a, Vu Van Tien1,2,a, Le Thi Mai Huong1, Nguyen Trung Anh1, Dinh Thi Mai Thu1, Nguyen Thi Hoa1, Tong Thi Huong1, Dinh Thi Thu Ngan1, Pham Xuan Hoi1, Jae-Yean Kim2*, Nguyen Van Dong1* 1National Key Laboratory for Plant Cell Biotechnology, Agricultural Genetics Institute, Vietnam Academy of Agricultural Sciences, Hanoi, Vietnam 2Division of Applied Life Science (BK21 program), Plant Molecular Biology and Biotechnology Research Center, Gyeongsang National University, Republic of Korea. aEqually contributed *Email: kimjaeyean@gmail.com and dongjircas@yahoo.com Summary Soybean (Glycine max L.) is one of the most highly valuable legumes planted in many regions around the world. However, soybean is considered one of the most drought susceptible plants. The NAC transcription factors have been well-known to be involved in regulation of plant response and tolerance to drought. GmNAC29 was shown to act as a negative regulator in plant response to abiotic stresses, including drought. Thus, in this study, we conducted to construct the CRISPR/Cas9 gene-editing vector system carrying sgRNAs of GmNAC29. The result will provide a CRISPR/Cas9-mediated mutagenic system for the GmNAC29 gene related to drought tolerance in soybean plants. Keywords: CRISPR/Cas9, drought stress, NAC, PCR, soybean. Người phản biện: TS. Nguyễn Xuân Thắng Ngày nhận bài: 13/7/2020 Ngày thông qua phản biện: 13/8/2020 Ngày duyệt đăng: 20/8/2020
File đính kèm:
 thiet_ke_he_thong_cau_truc_vector_crisprcas9_de_chinh_sua_ge.pdf
thiet_ke_he_thong_cau_truc_vector_crisprcas9_de_chinh_sua_ge.pdf

