Phân tích sự đa hình cây xoài (Mangifera indica L.) bằng kỹ thuật SSR tại đồng bằng sông Cửu Long
Chỉ thị phân tử SSR đã được sử dụng để nghiên cứu sự khác biệt di truyền với 30 giống xoài khác nhau. Trong số 66 mồi SSR, 31 mồi được sao chép với tổng số là 1.178 băng trong đó có 130 alen. Số lượng trung bình các mảnh gen được khuếch đại bằng marker SSR (kích thước 130 bp đến 400 bp). Primer SSR được sử dụng trong nghiên cứu, sản phẩm sản xuất PCR cho tỷ lệ biến động từ 90% tới 100%, hệ số PIC biến động từ 0,17 đến 0,85. Kết quả thu được chỉ số đa dạng Shannon I (5.312), sự đa dạng di truyền/locus – H (= 0,351) và hiệu quả allele/locus - AEP (10,9 1-11,35. Kết quả nghiên cứu này làm cơ sở di truyền để nghiên cứu trên các giống xoài khác nhau tại ĐBSCL. Băng điện di được phân tích bằng phương pháp UPMGA chia thành 4 nhóm chính có mối liên quan di truyền, trong đó đặc biệt chú ý đến chương trình nhân giống hiệu quả với mục tiêu cải tiến giống. Dựa vào số lượng và tần số ghi điểm của các đoạn DNA, tỷ lệ đa hình và các thông số hiệu quả khác sau khi tổng hợp lại thì dường như MiIIHR_i929, MiKVR_m056, LangXoai F4-R4 và Lang Xoài F2-R2 là những marker có hiệu quả và có thể được sử dụng để sàng lọc phân tử trong nguồn gene cây xoài cho đa hình và nhiều alen. Những phát hiện của nghiên cứu này tạo điều kiện cải thiện quản lý bảo tồn của nguồn gen và giúp cho các nghiên cứu chọn giống trong tương lai

Trang 1

Trang 2
Trang 3

Trang 4
Trang 5
Trang 6
Trang 7

Trang 8

Trang 9

Trang 10
Tóm tắt nội dung tài liệu: Phân tích sự đa hình cây xoài (Mangifera indica L.) bằng kỹ thuật SSR tại đồng bằng sông Cửu Long

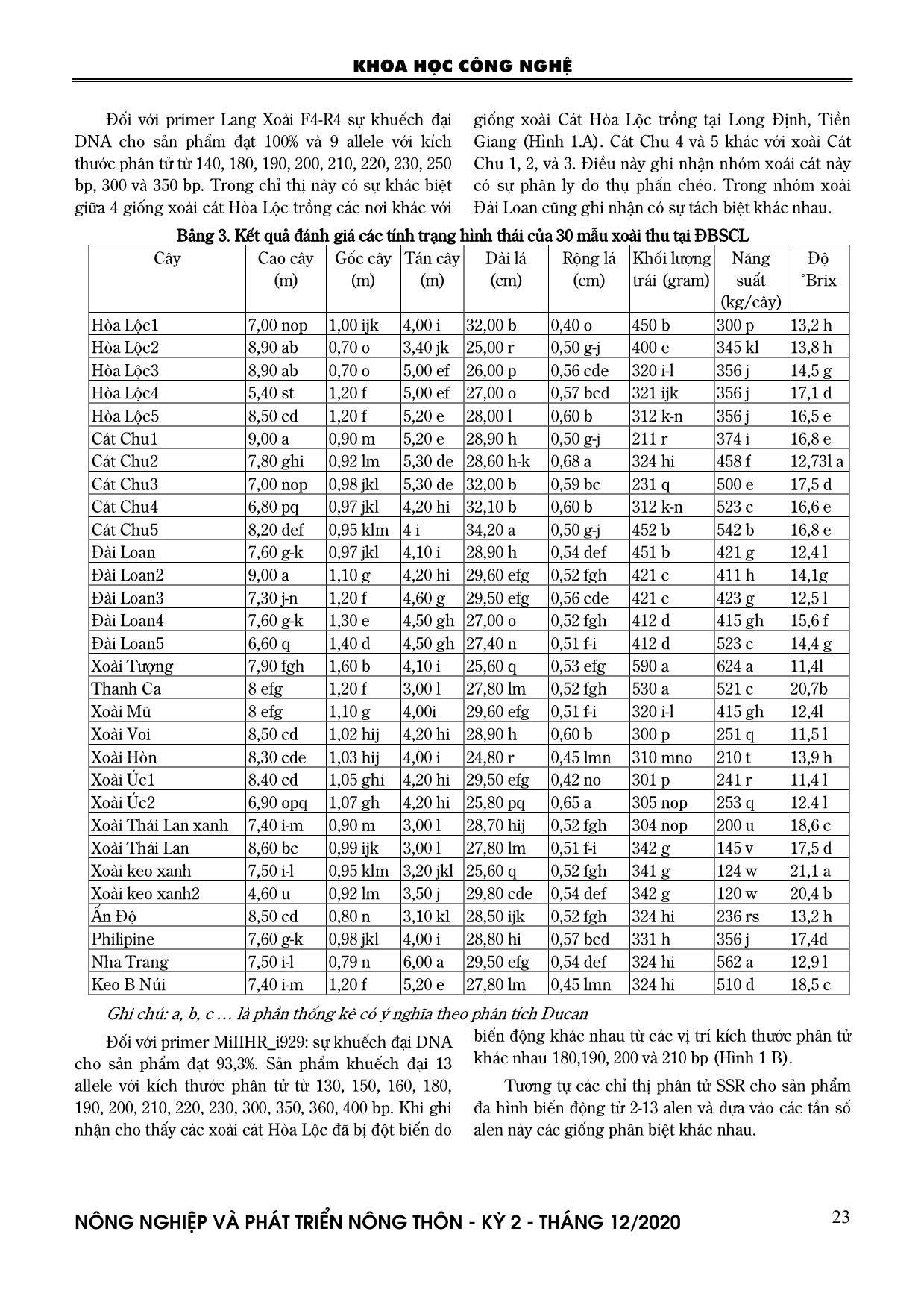
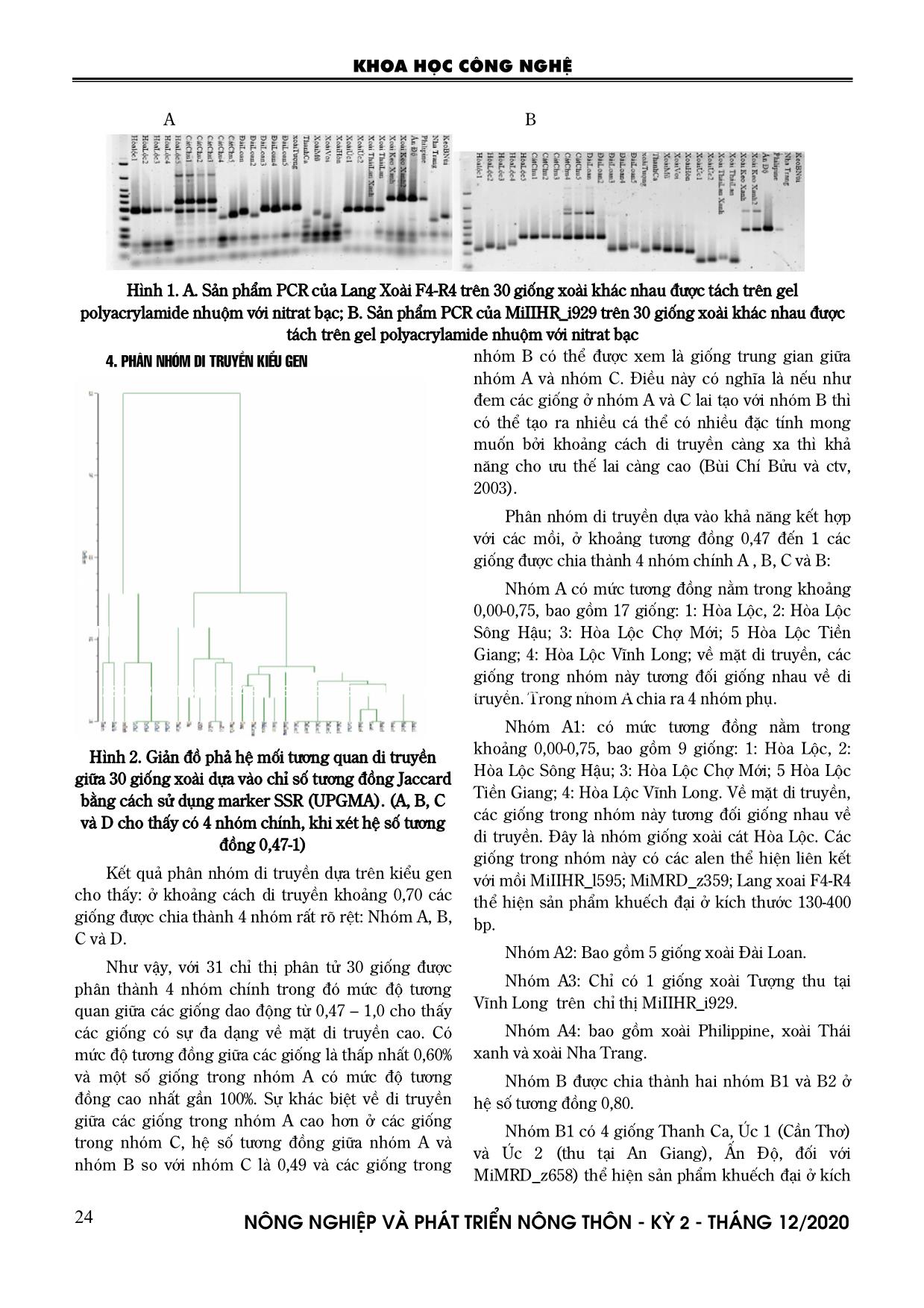
ha Trang 7,50 i-l 0,79 n 6,00 a 29,50 efg 0,54 def 324 hi 562 a 12,9 l Keo B Núi 7,40 i-m 1,20 f 5,20 e 27,80 lm 0,45 lmn 324 hi 510 d 18,5 c Ghi chú: a, b, c là phần thống kê có ý nghĩa theo phân tích Ducan Đối với primer MiIIHR_i929: sự khuếch đại DNA cho sản phẩm đạt 93,3%. Sản phẩm khuếch đại 13 allele với kích thước phân tử từ 130, 150, 160, 180, 190, 200, 210, 220, 230, 300, 350, 360, 400 bp. Khi ghi nhận cho thấy các xoài cát Hòa Lộc đã bị đột biến do biến động khác nhau từ các vị trí kích thước phân tử khác nhau 180,190, 200 và 210 bp (Hình 1 B). Tương tự các chỉ thị phân tử SSR cho sản phẩm đa hình biến động từ 2-13 alen và dựa vào các tần số alen này các giống phân biệt khác nhau. M KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 2 - TH¸NG 12/2020 24 A B Hình 1. A. Sản phẩm PCR của Lang Xoài F4-R4 trên 30 giống xoài khác nhau được tách trên gel polyacrylamide nhuộm với nitrat bạc; B. Sản phẩm PCR của MiIIHR_i929 trên 30 giống xoài khác nhau được tách trên gel polyacrylamide nhuộm với nitrat bạc 4. PHÂN NHÓM DI TRUYỀN KIỂU GEN Hình 2. Giản đồ phả hệ mối tương quan di truyền giữa 30 giống xoài dựa vào chỉ số tương đồng Jaccard bằng cách sử dụng marker SSR (UPGMA). (A, B, C và D cho thấy có 4 nhóm chính, khi xét hệ số tương đồng 0,47-1) Kết quả phân nhóm di truyền dựa trên kiểu gen cho thấy: ở khoảng cách di truyền khoảng 0,70 các giống được chia thành 4 nhóm rất rõ rệt: Nhóm A, B, C và D. Như vậy, với 31 chỉ thị phân tử 30 giống được phân thành 4 nhóm chính trong đó mức độ tương quan giữa các giống dao động từ 0,47 – 1,0 cho thấy các giống có sự đa dạng về mặt di truyền cao. Có mức độ tương đồng giữa các giống là thấp nhất 0,60% và một số giống trong nhóm A có mức độ tương đồng cao nhất gần 100%. Sự khác biệt về di truyền giữa các giống trong nhóm A cao hơn ở các giống trong nhóm C, hệ số tương đồng giữa nhóm A và nhóm B so với nhóm C là 0,49 và các giống trong nhóm B có thể được xem là giống trung gian giữa nhóm A và nhóm C. Điều này có nghĩa là nếu như đem các giống ở nhóm A và C lai tạo với nhóm B thì có thể tạo ra nhiều cá thể có nhiều đặc tính mong muốn bởi khoảng cách di truyền càng xa thì khả năng cho ưu thế lai càng cao (Bùi Chí Bửu và ctv, 2003). Phân nhóm di truyền dựa vào khả năng kết hợp với các mồi, ở khoảng tương đồng 0,47 đến 1 các giống được chia thành 4 nhóm chính A , B, C và B: Nhóm A có mức tương đồng nằm trong khoảng 0,00-0,75, bao gồm 17 giống: 1: Hòa Lộc, 2: Hòa Lộc Sông Hậu; 3: Hòa Lộc Chợ Mới; 5 Hòa Lộc Tiền Giang; 4: Hòa Lộc Vĩnh Long; về mặt di truyền, các giống trong nhóm này tương đối giống nhau về di truyền. Trong nhóm A chia ra 4 nhóm phụ. Nhóm A1: có mức tương đồng nằm trong khoảng 0,00-0,75, bao gồm 9 giống: 1: Hòa Lộc, 2: Hòa Lộc Sông Hậu; 3: Hòa Lộc Chợ Mới; 5 Hòa Lộc Tiền Giang; 4: Hòa Lộc Vĩnh Long. Về mặt di truyền, các giống trong nhóm này tương đối giống nhau về di truyền. Đây là nhóm giống xoài cát Hòa Lộc. Các giống trong nhóm này có các alen thể hiện liên kết với mồi MiIIHR_l595; MiMRD_z359; Lang xoai F4-R4 thể hiện sản phẩm khuếch đại ở kích thước 130-400 bp. Nhóm A2: Bao gồm 5 giống xoài Đài Loan. Nhóm A3: Chỉ có 1 giống xoài Tượng thu tại Vĩnh Long trên chỉ thị MiIIHR_i929. Nhóm A4: bao gồm xoài Philippine, xoài Thái xanh và xoài Nha Trang. Nhóm B được chia thành hai nhóm B1 và B2 ở hệ số tương đồng 0,80. Nhóm B1 có 4 giống Thanh Ca, Úc 1 (Cần Thơ) và Úc 2 (thu tại An Giang), Ấn Độ, đối với MiMRD_z658) thể hiện sản phẩm khuếch đại ở kích 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 M M M KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 2 - TH¸NG 12/2020 25 thước 170-200 bp), MRD_z414 (thể hiện sản phẩm khuếch đại ở kích thước 170- 220 bp). Nhóm B2 bao gồm các giống: gồm 1 giống xoài Thái Lan thể hiện sản phẩm khuếch đại ở kích thước MiIIHR_i929: Sản phẩm khuếch đại 13 allele với kích thước phân tử từ 130- 400 bp. Vị trí phân biệt được alen của B2 nằm vị trí 130 bp. Nhóm C: Chia ra hai nhóm: Nhóm C1: Có 4 giống Cát Chu. Nhóm này hầu hết đa hình trên chỉ thị MiIIHR_i929 (với 13 alen với kích thước phân tử từ 130, 150, 160, 180,190, 200, 210, 220, 230, 300, 350, 360, 400 bp). Tuy nhiên với nhóm xoài Cát Chu nằm vị trí alen với kích thước phân tử là 220, 230, 300, 350, 360, 400bp; MiMRD_z359 với chỉ thị phân tử 200-400 bp theo thứ tự. Nhóm C2: Có 1 giống Keo xanh thể hiện trên phân tử MiIIHR_i929, với alen từ 130 - 220 bp. Nhóm D: Có hai giống: Xoài Voi và xoài mủ. Nhóm này hầu hết đa hình trên chỉ thị MiMRD_m660 với chỉ thị phân tử RM 250- 290 bp và LangXoai2 F3-R3 với kích thước phân tử 190-230 bp. 5. THẢO LUẬN Việc xác định thay đổi chỉ dựa trên kiểu hình đặc điểm là không chính xác do ảnh hưởng của môi trường và số giới hạn của các đặc điểm của các giống. Điều này phù hợp với Hameedunnisa và ctv, 2014. Các kiểu gen được sử dụng trong nghiên cứu này cho thấy mức độ cao của dị hợp tử quan sát khác nhau, từ 0,07 đến 0,56. Đặc biệt phân biệt được dị hợp tử trên xoài cát Hòa Lộc trên 5 vùng khác nhau. Trong số 31 chỉ thị phân tử SSR, với 30 giống xoài (Hình 3) hiển thị các vạch băng của các cây một cách đồng nhất. Các chỉ thị phân tử SSR có tổng số 130 alen thể hiện tính đa hình. Trung bình số đoạn DNA được khuếch đại bằng marker SSR trong nghiên cứu này là dao động từ 2 đến 13 alen (kích thước từ 130-400 bp). Với 31 chỉ thị có một sự biến đổi cao của các đoạn DNA được tạo ra từ marker SSR có thể là do sự khác biệt trong vị trí gắn trên toàn bộ các alen của các giống xoài khác nhau. Tính hiệu quả của các chỉ thị phân tử sử dụng trong SSR là xác định được số lượng bằng cách ước đoán dựa vào sự khác biệt của các thông số di truyền (Bảng 1). Số lượng alen trên mỗi locus dao động từ 2 tại 3 của locus (MiKVR_a394, MiKVR_b283) đến 7 tại 1 của locus (MiMRD_z359), 9 alen (Lang Xoai F-R) và 13 locus (MiIIHR_i929) với giá trị trung bình là 4,1 alen (Bảng 1). Với 130 vị trí có thể được phân biệt và phân loại thành 30 kiểu gen đã tìm ra đa dạng di truyền phân tử dựa vào chỉ số Shannon trên từng chỉ thị phân tử trung bình là 5,789 với giá trị dao động từ 10,91-11,35 cho thấy đa dạng di truyền rộng. Khi so sánh các giá trị PIC, đa dạng di truyền (Hep) là dạng đa dạng có thể xác định của bốn nhóm giống, (0,504 và 0,337). Các giá trị này tương tự phân tích của Sagar S. và ctv, 2007) biểu hiện tính hiệu quả của thông tin loci SSR trong nghiên cứu này cũng ghi nhận sự đa hình cao trên chỉ thị phân tử như MiMRD_z359. Trong nghiên cứu này, Hep (chỉ số đa dạng di truyền) cho thấy sự đồng nhất đáng chú ý trên từng chỉ thị phân tử và dao động từ 0,72% (MiIIHR_i929). Hơn nữa, Hep cho thấy mối liên hệ tích cực và có ý nghĩa (r = 0,997, P < 0,001) với ảnh hưởng alen/locus (10,91-11,35). Thông số của chỉ thị phân tử đã được tính toán để phát hiện các đặc tính của từng cá thể bằng chỉ thị phân tử riêng biệt để xác định các locus đa hình trên các loại giống xoài khác nhau. Băng hình điện di được phân tích bằng phương pháp UPGMA (Hình 2) cho thấy quá trình phân nhóm kiểu gene thành 4 nhóm chính. Có một mối liên hệ chặt chẽ giữa các kiểu gene đã được ghi nhận. Mối tương quan di truyền như thế là rất có ý nghĩa, nó cung cấp cho các vật liệu tốt cho nghiên cứu cho chương trình lai giống xoài sau này. Bên cạnh đó, sự hiểu biết về sự đa dạng của nguồn gen của các giống để khai thác dị hợp tử bằng phân tích đa dạng. Ngoài ra việc phát hiện trong các nguồn gen cũng có thể ngăn chặn các giống lai có sự trùng lặp các alen, tránh nhầm lẫn trong di truyền mà có thể được gây ra cận giao khi lai. 6. KẾT LUẬN Phân tích kiểu hình của 30 cây xoài dựa vào năng suất và hình thái đã phân biệt được các giống xoài khác nhau. Tuy nhiên sự biến động của các giống rất lớn. Điều này còn tùy thuộc vào giống, môi trường, tuổi của cây trồng. Phân tích trên chỉ thị phân tử của SSR: trong tổng số 66 primers tiến hành nghiên cứu đa dạng di truyền thì chỉ có 31 chỉ thị phân tử cho sản phẩm khuếch đại trên 30 giống xoài. Trung bình số đoạn DNA được khuếch đại bằng marker SSR trong nghiên cứu này là dao động từ 2 đến 13 alen (kích thước phân tử biến động từ 130-400 bp). Thông qua các dữ liệu chỉ thị phân tử SSR với 31 chỉ thị phân tử được sử dụng, 30 giống được phân thành 4 nhóm chính. KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 2 - TH¸NG 12/2020 26 Bản đồ phân nhóm dựa phương pháp chỉ thị phân tử cũng được thiết lập với bốn nhóm khác nhau. Trong đó đáng chú ý có bốn chỉ thị MiIIHR_i929; MiKVR_m056, Lang Xoai F4-R4 và LangXoaiF2-R2. Có thể phân biệt độ thuần của các giống xoài khác nhau. Chỉ số đa dạng của cây xoài phân tích theo phương pháp SSR cao (H = 0,315). Các kết quả được trình bày ở đây là những bước đầu tiên hướng tới một hiểu biết tốt hơn về các giống cây xoài tại ĐBSCL và có thể phục vụ cho các nghiên cứu lai tạo loại cây này trong tương lai. LỜI CẢM ƠN Tác giả chân thành cảm ơn Sở Khoa học và Công nghệ Cần Thơ đã cấp kinh phí để thực hiện đề tài này. TÀI LIỆU THAM KHẢO 1. Bùi Chí Bửu, Phạm Thị Thu Hà, Đòn Văn Hòn, Bùi Phước Tâm, Châu Thanh Nhã, Nguyễn Thị Lang, 2013. Nghiên cứu biến động di truyền tính chịu nóng trên quần thể hồi giao cây lúa (Oryza sativa L.). Tạp chí Nông nghiệp và PTNT, số 2/2013 trang 10-15. 2. Bui Chi Buu, Nguyen Thi Lang, 2003. Application of molecular marker to rice breeding in mekong delta Vietnam. Agriculture rural development. 3. Hameedunnisa Begum, Medagam Thirupathi Reddy, Surapaneni Malathi, Boreddy Purushotham Reddy, Gonela, Narshimulu, Javaregowda Nagaraju and Ebrahimali Abubaker Siddiq, 2014. Morphological and Microsatellite Analysis of Intravarietal Variability in 'Cherukurasam' Cultivar of Mango (Mangifera indica L.). 2014. Jordan Journal of Agricultural Sciences. Volume 10, No.3 2014. 4. Kundapura V Ravishankar; M R DINESH, P. Nischita, B. S. Sandya, 2015. Development and characterization of microsatellite markers in mango (Mangifera indica) using next-generation sequencing technology and their transferability across species. Molecular Breeding 35(3).DOI: 10.1007/s11032-015- 0289-2. 5. Masroor HM, Anjum MA, Hussain S., Ejaz S., Ahmad S., Ercisli S., Zia-Ul-Haq M. Zinc improves yields and the quality of the mango is cultivated in the limestone soil. Erwerbs-Obstbau, 2016; 58:49 Bóng55. DOI: 10.1007/s10341-015-0258-2. [CrossRef] [Google scholar. 6. Munir T., Perkins M., Kaing E., Strack M., 2015. Carbon dioxide and the main production of a bog treed boreal: Reaction to warming and simulation lowering the water level of climate change. Biochemistry. 2015; 12:1091 from 1111. DOI: 10.5194/BG-12-1091-2015. [CrossRef] [Google scholar]. 7. Mukherjee, S. K., 1950. Mango: its allopolyploid nature. Nature, 166: 196– 7. 8. Mukherjee, S. K., 1951. Origin of Mango. Indian J. Genet. Pl. Breed., 11:49–56. 9. Mukherjee, S. K., 1957. Cytology of some Malayan Species of Mangifera. Cytologia, 22: 239–41. 10. Martynov SP, Dobrotvorskaya TV, Dotlacil L, Stehno Z, Faberova I, Bares I., 2003. Genealogical approach to the formation of the winter wheat core collection. Russian J Genet 2003; 39: 917-923. 11. Nguyễn Thị Lang, 2002. Những phương pháp cơ bản trong công nghệ sinh học. Nhà xuất bản Nông nghiệp. 12. Nguyen Thi Lang, Bui Phuoc Tam, Nguyen Van Hieu, Chau Thanh Nha, Abdelbagi Ismail, Russell Reinke and Bui Chi Buu, 2014. Evaluation of rice landraces in Vietnam using SSR markers and morphological characters. Sabrao Journal of Breeding and Genetics, 46(1) p1-20. 13. Nguyễn Thị Lang, 2009. Ứng dụng chỉ thị phân tử PCR trong chọn dòng thuần cây xoài cát sông Hậu. Báo cáo tổng kết đề tài cấp TP. Cần Thơ. 14. IPGRI, 2006. Descriptors for mango (Mangifera indica L). International Plant Genetic. Resources Institute, Rome, Italy. 15. Roy, B. and S. S. Visweswariya, 1951. Cytogenetics of Mango and Banana. 16. Rohlf, F. J., 2000. NTSYS-pc: Numerical taxonomy and multivariate analysis system, version 2.1 Exeter Software, Setauket, New York, USA. 17. Sagar S. Pandit, Sirsha Mitra, Ashok P. Giri, Keshav H. Pujari, Bhimarao P. Patil, Narayan D. Jambhale and Vidya S. Gupta., 2007. Genetic diversity analysis of mango cultivars using inter KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 2 - TH¸NG 12/2020 27 simple sequence repeat markers. CURRENT SCIENCE, VOL. 93, NO. 8, 25 OCTOBER 2007. Sagar S. Pandit, Sirsha Mitra, Ashok P. Giri, Keshav H. Pujari, Bhimarao P. Patil, Narayan D. Jambhale and Vidya S. Gupta1.2014. Genetic diversity analysis of mango cultivars using inter simple sequence repeat markers. https://www.researchgate.net/publication/22869482 2. 18. UNCTAD. Mango, 2016. An Infocomm commodity record. United Nations; New York, NY, United States: Geneva, Switzerland. 2016. Ward 21. [Google scholar). Weir BS. Genetic data analysis II: Methods for discrete population genetic data. Sinauer Publishers, Sunderland, MA, USA; 1996. Nguồn https://www.ibm.com/support/pages/downloading- ibm-spss-statistics-20. GENETIC DIVERSITY STUDIES MANGO (Mangifera indica L.) BY SIMPLE SEQUENCE REPEAT MARKERS (SSR) Nguyen Thi Lang1, Nguyen Thi Khanh Tran1, Bui Chi Cong1, Bui Chi Hieu1, Bui Chi Buu1 1 High Agricultural Technology Research Institute for Mekong delta Summary SSR markers researh for have been used to genetic distinctiveness among 30 mango varieties. Out of the 66 primers for SSR 31 primers reproducte a total 1,178 bands of which 130 alen were polymorphic. The average number of fragment amplified by SSR marker across the mango have been 130-400 bp. SSR markers across that from 90-100% polymorphism. Information contents: PIC (0.17 to 0.85), genetice diversity H (= 0.351) and allele/locus - AEP (10.91-11.35 Shannon I (5,312). Band analysied by UPMGA (Unweighted pair group method with arithmetic Mean) showed four major clusters and close relatedress amond 30 varieties. This is maybe significant for designing breeding program towards. Some markers good for efficient breeding such as MiIIHR_i929; MiKVR_m056, Lang Xoai F4-R4 and LangXoaiF2-R2. The analysis of the population structure of the individuals also showed four subpopulations, which supported the generated dendrogram. The findings of this study facilitate improved conservation management of the germplasm and help to find strategies for future breeding activities. Keywords: Multi-image analysis, SSR markers, mango varieties, primer, genetic parameters. Người phản biện: GS.TS. Ngô Xuân Bình Ngày nhận bài: 31/7/2020 Ngày thông qua phản biện: 31/8/2020 Ngày duyệt đăng: 7/9/2020
File đính kèm:
 phan_tich_su_da_hinh_cay_xoai_mangifera_indica_l_bang_ky_thu.pdf
phan_tich_su_da_hinh_cay_xoai_mangifera_indica_l_bang_ky_thu.pdf

