Nghiên cứu đa dạng di truyền và nhận dạng một số giống quýt bản địa của Việt Nam dựa trên trình tự ITS hệ gen nhân
Nguồn gen quýt (C. reticulata) bản địa của Việt Nam rất đa dạng và phong phú, cần được khai thác và sử dụng, phục vụ cho phát triển kinh tế, xã hội một cách hiệu quả và bền vững. Mười bốn mẫu/giống quýt được thu thập ở 10 tỉnh/thành khác nhau đã được giải trình tự nhờ khuếch đại locus ITS trong hệ gen nhân. Đoạn trình tự ITS của 14 mẫu/giống quýt đã được so sánh trình tự với gen tham chiếu công bố trên NCBI. Kết quả so sánh cho thấy: độ tương đồng về trình tự nucleotide của các mẫu/giống quýt nghiên cứu và trình tự tham chiếu đã công bố trên NCBI dao động từ 94,0% đến 99,59% với độ bao phủ dao động từ 96-97%. Hệ số tương đồng di truyền của 14 mẫu/giống quýt nghiên cứu dao động từ 93,59 - 99,73%, 14 mẫu/giống quýt nghiên cứu được phân thành 4 nhóm chính. Dựa vào sự khác biệt nucleotide ở một số vị trí trong trình tự vùng ITS, có thể nhận dạng chính xác sáu mẫu/giống quýt đó là quýt Tích Giang (Q4), quýt Hương Cần (Q5), quýt đỏ Ngọc Hội (Q9), quýt Hôi (Q11), quýt ngọt Hà Giang (Q12) và quýt Bộp (Q13). Kết quả này rất có ý nghĩa, phục vụ cho công tác bảo tồn, khai thác và sử dụng có hiệu quả nguồn gen quýt bản địa của Việt Nam

Trang 1

Trang 2

Trang 3

Trang 4
Trang 5

Trang 6

Trang 7
Tóm tắt nội dung tài liệu: Nghiên cứu đa dạng di truyền và nhận dạng một số giống quýt bản địa của Việt Nam dựa trên trình tự ITS hệ gen nhân

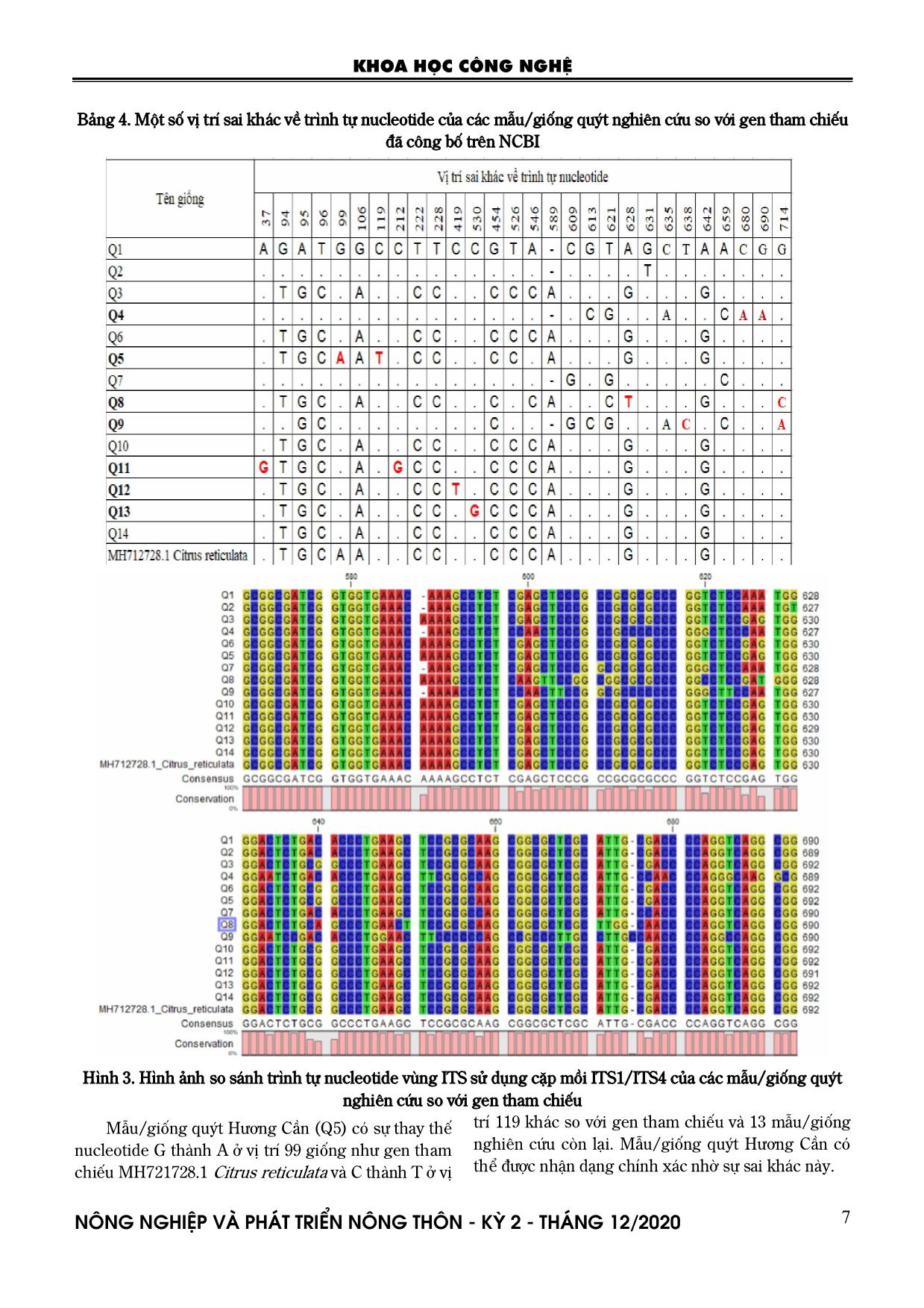
Bắc Kạn Ba Bể, Bắc Kạn Q14 - Cặp mồi ITS1/ITS4 (TCCGTAGGTGAACCTGCGG/TCCTCCGCTTATT GATATGC) (Vilgalys R. et al., 1994). 2.2. Phương pháp nghiên cứu - Mẫu lá được thu thập và tách chiết ADN tổng số theo phương pháp CTAB của Doyle JJ at el. (1990) có cải tiến và kiểm tra nồng độ bằng phương pháp điện di gel agarose 1%. - Phản ứng PCR được tiến hành trên máy Veriti 96 well Thermal cycler. Tổng thể tích phản ứng là 15 µl, bao gồm: 5 µl ADN; 0,15 µM mồi; 0,2 mM dNTPs; 1X Buffer PCR; 2,5 mM MgCl2 và 0,25 U Taq polymerase. Chu trình nhiệt phản ứng PCR gồm: 94C (5 phút), 36 chu kỳ [94C (1 phút), 56C (45 giây), 72C (50 giây)] và kết thúc ở 72C (7 phút), giữ ở 4C. - Sản phẩm PCR được điện di trên gel agarose 1,5% sử dụng đệm TAE. Gel được nhuộm ethidium bromide 0,5 mg/ml và soi trên máy Alpha Imager 1220 (Alpha Innotech, CA, USA). - Sản phẩm PCR được thôi gel theo qui trình hướng dẫn của bộ kit Qiagen; - Giải trình tự: Sản phẩm PCR sau khi được tinh sạch, được giải trình tự tại Công ty Apical Scientific (Malaysia). - Phương pháp xử lí số liệu: Kết quả giải trình tự được so sánh với trình tự tham chiếu trên NCBI và được phân tích bằng chương trình MEGA v6.06. 3. KẾT QUẢ NGHIÊN CỨU 3.1. Kết quả giải trình tự vùng ITS Hình 1. Ảnh điện di sản phẩm PCR của 14 mẫu/giống quýt nghiên cứu với cặp mồi ITS1/ITS4 Q1-Q14: Các mẫu/giống quýt bản địa ở bảng 1; M: Marker generuler 100 bp plus DNA Các mẫu/giống quýt được thu thập tại các tỉnh/thành: Đồng Tháp, Thừa Thiên - Huế, Hà Giang, Bắc Kạn, Yên Bái, Phú Thọ, Tuyên Quang, 1.000 bp 500 bp bp o bp KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 2 - TH¸NG 12/2020 5 Sơn La, Lạng Sơn và Hà Nội được tách chiết ADN và đưa về nồng độ 50 ng/µl để tiến hành thực hiện phản ứng PCR với cặp mồi ITS1/ITS4. Kết quả điện di sản phẩm PCR cho thấy các mẫu quýt nghiên cứu đều cho băng đơn hình với kích thước khoảng 750 bp (Hình 1). Bảng 2. Đánh giá mức độ tương đồng và độ bao phủ về trình tự DNA vùng ITS của các mẫu/giống quýt nghiên cứu so với gen tham chiếu đã công bố trên NCBI TT Tên giống Kí hiệu Trình tự tham chiếu Độ tương đồng (%) Độ bao phủ (%) 1 Quýt đường Q1 MH712728.1_Citrus_reticulata 97,10 97,00 2 Quýt Chiềng Cọ Q2 MH712728.1_Citrus_reticulata 96,96 97,00 3 Quýt hồng (Tiều Sơn) Q3 MH712728.1_Citrus_reticulata 99,59 97,00 4 Quýt Tích Giang Q4 MH712728.1_Citrus_reticulata 95,03 97,00 5 Quýt Hương Cần Q5 MH712728.1_Citrus_reticulata 99,45 97,00 6 Quýt Tràng Định Q6 MH712728.1_Citrus_reticulata 99,59 97,00 7 Quýt lửa Q7 MH712728.1_Citrus_reticulata 96,55 97,00 8 Quýt Đông Khê Q8 MH712728.1_Citrus_reticulata 96,44 97,00 9 Quýt đỏ Ngọc Hội Q9 MH712728.1_Citrus_reticulata 94,00 96,00 10 Quýt Chum Q10 MH712728.1_Citrus_reticulata 99,59 97,00 11 Quýt Hôi Q11 MH712728.1_Citrus_reticulata 97,41 97,00 12 Quýt ngọt Hà Giang Q12 MH712728.1_Citrus_reticulata 99,31 97,00 13 Quýt Bộp Q13 MH712728.1_Citrus_reticulata 99,45 97,00 14 Quýt Bắc Kạn Q14 MH712728.1_Citrus_reticulata 99,59 97,00 Các đoạn trình tự ITS của 14 mẫu/giống quýt nghiên cứu được so sánh mức độ tương đồng với các trình tự tham chiếu đã công bố trên NCBI sử dụng chương trình BLAST. Kết quả phân tích cho thấy độ tương đồng về trình tự của các mẫu/giống quýt so với gen tham chiếu dao động từ 94,00% đến 99,59% với độ bao phủ dao động từ 96 - 97%. Trong đó mẫu/giống quýt đỏ Ngọc Hội có mức độ tương đồng về trình tự so với gen tham chiếu thấp nhất là 94%. Các mẫu/giống quýt hồng (Tiểu Sơn), quýt Tràng Định, quýt Chum và quýt Bắc Kạn có mức độ tương đồng về trình tự so với gen tham chiếu cao nhất là 99,59%. Điều này cho thấy các trình tự vùng ITS khuếch đại của một số mẫu/giống quýt nghiên cứu có mức độ tương đồng cao so với trình tự đã được công bố trên NCBI. 3.2. Kết quả phân tích đa dạng di truyền các mẫu/giống quýt nghiên cứu Kết quả phân tích các trình tự thông qua phần mềm MEGA v6.06 thu được số liệu ở bảng 3 cho thấy có hệ số tương đồng di truyền của 14 mẫu/giống quýt nghiên cứu dao động từ 93,59 - 99,73%. Hệ số tương đồng di truyền thấp nhất là 93,59% giữa mẫu/giống Q3 và Q9. Hệ số tương đồng di truyền cao nhất là 99,73% giữa mẫu/giống Q3 và Q10, giữa các mẫu/giống Q10 - Q13 - Q14. Dựa vào kết quả phân tích trình tự DNA vùng ITS cho thấy 14 mẫu/giống quýt nghiên cứu được phân thành 4 nhóm (Hình 2, bảng 3). Hình 2. Cây phát sinh chủng loại vùng gen ITS của 14 mẫu/giống quýt nghiên cứu và gen tham chiếu KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 2 - TH¸NG 12/2020 6 Bảng 3. Hệ số tương đồng di truyền giữa 14 mẫu/giống quýt nghiên cứu và gen tham chiếu Q1 Q2 Q3 Q4 Q5 Q6 Q7 Q8 Q9 Q10 Q11 Q12 Q13 Q14 MH7 Q1 Q2 99,18 Q3 96,86 96,86 Q4 97,26 97,26 95,09 Q5 97,13 97,13 99,45 95,08 Q6 96,59 96,86 99,32 94,82 99,05 Q7 99,04 98,77 96,59 97,67 96,58 96,45 Q8 95,08 95,35 96,73 93,97 96,99 96,58 95,21 Q9 94,79 95,07 93,59 96,02 93,85 93,72 95,61 93,83 Q10 96,86 97,14 99,73 95,09 99,45 99,45 96,45 96,86 93,72 Q11 96,72 96,99 99,32 94,95 99,32 99,05 96,31 96,72 93,58 99,59 Q12 96,72 96,99 99,45 94,95 99,18 99,32 96,58 96,99 93,84 99,59 99,18 Q13 96,59 96,86 99,59 94,82 99,32 99,45 96,45 96,86 93,72 99,73 99,32 99,59 Q14 96,59 96,86 99,59 94,82 99,32 99,45 96,45 96,86 93,72 99,73 99,32 99,59 99,73 MH7 95,09 94,95 97,68 93,04 97,54 97,68 94,67 94,95 91,8 97,68 97,27 97,54 97,68 97,81 Nhóm thứ nhất (nhóm I) gồm một mẫu duy nhất là giống quýt đỏ Ngọc Hội (Q9). Dựa vào trình tự nucleotide cũng cho thấy mẫu/giống Q9 có sự đa hình nucleotide so với gen tham chiếu; mẫu/giống Q9 có hệ số tương đồng di truyền so với gen tham chiếu là 91,80%. Nhóm thứ hai (nhóm II) gồm 8 mẫu/giống quýt là Q3, Q5, Q6, Q10, Q11, Q12, Q13, Q14 và gen tham chiếu MH721728.1 Citrus reticulata. Các mẫu/giống trong nhóm I có hệ số tương đồng di truyền cao dao động từ 99,05% (mẫu/giống Q6 với Q5 và Q11) đến 99,73% (mẫu/giống Q3 và Q10, các cặp mẫu/giống Q10 - Q13 - Q14). Hệ số tương đồng di truyền giữa các mẫu/giống quýt trong nhóm với mẫu tham chiếu MH712728.1_Citrusreticulata dao động từ 97,27 % (Q11) đến 97,81% (Q14). Nhóm thứ ba (nhóm III) cũng có duy nhất giống quýt Đông Khê (Q8), mẫu/giống này có hệ số tương đồng di truyền so với các mẫu/giống thuộc nhóm khác dao động từ 93,83% (Q8-Q9) đến 96,99% (Q8- Q12, Q5-Q8) và với gen tham chiếu là 94,95%; Nhóm thứ tư (nhóm IV) gồm 4 mẫu/giống là Q1, Q2, Q4 và Q7. Các mẫu/giống trong nhóm IV có hệ số tương đồng di truyền cao dao động từ 97,26% (Q1-Q4, Q2-Q4) đến 99,18% (Q1-Q2) và với gen tham chiếu dao động từ 93,04% (Q4) đến 95,09% (Q1). 3.3. Kết quả nhận dạng một số mẫu/giống quýt nghiên cứu Kết quả so sánh trình tự của 14 mẫu/giống quýt nghiên cứu với trình tự gen tham chiếu đã công bố trên NCBI thông qua phần mềm MEGA v6.06 thu được ở hình 3 cho thấy: do có sự mất đoạn hay thêm đoạn ở một số vị trí trên đoạn gen nên số lượng nucleotide tổng số của mỗi đoạn trình tự khác nhau giữa 14 mẫu/giống quýt nghiên cứu. Chiều dài vùng ITS của 14 mẫu/giống quýt nghiên cứu dao động từ 719 nucleotide đến 734 nucleotide. Vùng ITS có sự đa hình trong trình tự nucleotide rất cao với 74 vị trí sai khác giữa các mẫu/giống nghiên cứu và gen tham chiếu. Trong đó, sáu mẫu/giống quýt Tích Giang (Q4), quýt Hương Cần (Q5), quýt đỏ Ngọc Hội (Q9), quýt Hôi (Q11), quýt ngọt Hà Giang (Q12) và quýt Bộp (Q13) có sự biến đổi nucleotide khác biệt so với các mẫu/giống còn lại và gen tham chiếu MH721728.1 Citrus reticulata (Bảng 4). Mẫu/giống quýt Tích Giang (Q4) có sự thay thế các nucleotide C, C, G thành A ở các vị trí 635, 680 và 690 tương ứng. Tuy nhiên ở 2 vị trí 680 và 690, chỉ có duy nhất mẫu/giống quýt Tích Giang có sự thay thế các nucleotide C, G thành A khác biệt so với gen tham chiếu và mẫu/giống khác trong tập đoàn nghiên cứu, vì vậy mẫu quýt này có thể được nhận dạng chính xác nhờ sự khác biệt này. KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 2 - TH¸NG 12/2020 7 Bảng 4. Một số vị trí sai khác về trình tự nucleotide của các mẫu/giống quýt nghiên cứu so với gen tham chiếu đã công bố trên NCBI Hình 3. Hình ảnh so sánh trình tự nucleotide vùng ITS sử dụng cặp mồi ITS1/ITS4 của các mẫu/giống quýt nghiên cứu so với gen tham chiếu Mẫu/giống quýt Hương Cần (Q5) có sự thay thế nucleotide G thành A ở vị trí 99 giống như gen tham chiếu MH721728.1 Citrus reticulata và C thành T ở vị trí 119 khác so với gen tham chiếu và 13 mẫu/giống nghiên cứu còn lại. Mẫu/giống quýt Hương Cần có thể được nhận dạng chính xác nhờ sự sai khác này. KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 2 - TH¸NG 12/2020 8 Mẫu/giống quýt Đông Khê (Q8) có thể được nhận dạng nhờ sự sai khác so với gen tham chiếu và mẫu/giống nghiên cứu ở vị trí 628 và 714. Ở hai vị trí này có sự thay thế các nucleotide A thành T và G thành C tương ứng. Mẫu/giống quýt đỏ Ngọc Hội (Q9) có sự thay thế các nucleotide T, G thành C và A ở vị trí 638 và 714 tương ứng. Vì vậy chỉ có thể nhận dạng chính xác mẫu/giống quýt đỏ Ngọc Hội nhờ sự khác biệt này so với các mẫu/giống khác trong tập đoàn nghiên cứu. Mẫu/giống quýt Hôi (Q11) có sự thay thế các nucleotide A, C thành G lần lượt ở 2 vị trí 37 và 212, khác biệt so với các mẫu/giống khác trong tập đoàn nghiên cứu, vì vậy có thể nhận dạng chính xác mẫu/giống quýt Hôi nhờ sự biến đổi nucleotide này. Mẫu/giống quýt ngọt Hà Giang (Q12) có sự thay thế nucleotide duy nhất ở vị trí 419, từ C thành T so với gen tham chiếu đã công bố trên NCBI và các mẫu/giống còn lại. Do đó mẫu/giống quýt ngọt Hà Giang có thể được nhận dạng chính xác nhờ sự sai khác này. Tương tự, mẫu/giống quýt Bộp (Q13) có sự thay thế nucleotide duy nhất ở vị trí 530, từ C thành G, khác so với các mẫu/giống còn lại và gen tham chiếu đã công bố trên NCBI. Vì vậy nhờ sự sai khác này nên mẫu/giống quýt Bộp có thể được nhận dạng chính xác trong tập đoàn nghiên cứu. 4. KẾT LUẬN Độ tương đồng về trình tự nucleotide của 14 mẫu/giống quýt nghiên cứu và trình tự tham chiếu đã công bố trên NCBI dao động từ 94,00% đến 99,59% với độ bao phủ dao động từ 96-97%. Các mẫu/giống quýt hồng (Tiểu Sơn), quýt Tràng Định, quýt Chum và quýt Bắc Kạn có mức độ tương đồng về trình tự so với gen tham chiếu cao nhất là 99,59%. Hệ số tương đồng di truyền của 14 mẫu/giống quýt nghiên cứu dao động từ 93,59 - 99,73%, 14 mẫu/giống quýt nghiên cứu được phân thành 4 nhóm. Dựa vào trình tự vùng ITS của 14 mẫu/giống quýt nghiên cứu có thể nhận dạng được sáu mẫu/giống quýt đó là: quýt Tích Giang (Q4), quýt Hương Cần (Q5), quýt đỏ Ngọc Hội (Q9), quýt Hôi (Q11), quýt ngọt Hà Giang (Q12) và quýt Bộp (Q13), do có sự thay thế nucleotide ở một số vị trí, khác biệt so với các mẫu/giống khác và gen tham chiếu MH721728.1 Citrus reticulata. Kết quả này rất có ý nghĩa, phục vụ cho công tác bảo tồn, khai thác và sử dụng có hiệu quả nguồn gen quýt bản địa của Việt Nam. LỜI CẢM ƠN Nghiên cứu này được thực hiện với sự hỗ trợ kinh phí của đề tài “Xây dựng cơ sở dữ liệu nguồn gen và mã vạch ADN (DNA barcode) cho các loài cây có múi (bưởi, cam và quýt) bản địa/địa phương của Việt Nam” thuộc Chương trình trọng điểm phát triển và ứng dụng công nghệ sinh học trong lĩnh vực nông nghiệp và phát triển nông thôn đến năm 2020, Bộ Nông nghiệp và PTNT. TÀI LIỆU THAM KHẢO 1. Doyle, J. J. and Doyle J. L. (1990). Isolation of plant DNA from fresh tissue. Focus, v.12:13-15. 2. Milena D. A., Marcia F. B. P., Frederique O.t, Ronan R., Edson M. A. S., Abelmon S. G., François L., Dominique G., Patrick O., and Fabienne M. (2019). Phylogenetic Origin of Primary and Secondary Metabolic Pathway Genes Revealed by C. maxima and C. reticulata Diagnostic SNPs. Front Plant Sci, 10: 1128. 3. Oueslati A., Salhi-Hannachi A., Luro F., Vignes H., Mournet P., Ollitrault P. (2017). Genotyping by sequencing reveals the interspecific C. Maxima/C. Reticulata admixture along the genomes of modern citrus varieties of Mandarins, Tangors, Tangelos, Orangelos and Grapefruits. Plos One, 12 (10): 1371. 4. Vilgalys R., Hopple J. S., and Hibbett D. S. (1994). Phylogenetic implications of generic concepts in fungal taxonomy: the impact of molecular systematic studies. Mycol. Helv. 6:73-91. 5. Wu G. A., Prochnik S., Jenkins J., Salse J., Hellsten U., Murat F., et al. (2014). Sequencing of diverse mandarin, pummelo and orange genomes reveals complex history of admixture during citrus domestication. Nat.Biotechnol. 32: 656-662. 6. Wu G. A., Terol J., Ibanez V., López-García A., Pérez-Román E., Borredá C., et al. (2018). Genomics of the origin and evolution of citrus. Nature, 554: 311- 316. 7.https://blast.ncbi.nlm.nih.gov/Blast.cgi?PRO GRAM=blastn&PAGE_TYPE=BlastSearch&BLAST_ SPEC=&LINK_LOC=blasttab&LAST_PAGE=tblastn 8. www.megasoftware.net KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 2 - TH¸NG 12/2020 9 STUDYING ON DIVERSITY AND IDENTIFICATION OF VIETNAMESE NATIVE Citrus reticulata SPECIES BASED ON RIBOSOMAL DNA INTENAL TRANSCRIBED SPACER SEQUENCE Nguyen Thuy Diep1, Le Thi Thu Trang2, Kieu Thi Dung1, Dang Thi Thanh Ha1, Tran Dang Khanh1, La Tuan Nghia2, Khuat Huu Trung1 1Agricultural Genetics Institue 2Plant Resources Center Summary The Vietnamese native of mandarin orange genetic sources (C. reticulata) is very diversity and plentiful, which should be exploited and used, service for effectively and sustainably socio-economic development. Fourteen mandarin orange landraces have been collected in 10 different provinces and sequenced by amplification of the ITS locus in the nuclear genome. The ITS sequences of 14 mandarin orange accessions have been compared with reference gene which published in NCBI. The results showed that: similarity of the nucleotide sequence of landraces and the published reference gene on NCBI ranged from 94.00% to 99.59% with coverage ranged from 96.00% to 97.00%. Genetic similarity coefficients of 14 mandarin orange accessions were in range from 93.59 to 99.73%, 14 mandarin orange accessions were divided into 4 main groups. Based on nucleotide differences in several positions in the ITS region sequence, six landraces have been identified correctly, include: Tich Giang (Q4), Huong Can (Q5) and Do Ngoc Hoi (Q9), Quyt Hoi (Q11), Ha Giang sweet tangerine (Q12) and Quyt Bop (Q13). This result is very useful for the conservation, exploitation and effective use of this precious genetic resources in Vietnam. Keywords: Nucleotide sequences, mandarin orange accessions, ITS, genetic diversity, PCR. Người phản biện: GS.TS. Ngô Xuân Bình Ngày nhận bài: 16/10/2020 Ngày thông qua phản biện: 16/11/2020 Ngày duyệt đăng: 23/11/2020
File đính kèm:
 nghien_cuu_da_dang_di_truyen_va_nhan_dang_mot_so_giong_quyt.pdf
nghien_cuu_da_dang_di_truyen_va_nhan_dang_mot_so_giong_quyt.pdf

