Đánh giá đa dạng di truyền trong các quần thể ba kích tím (Morinda officinalis F. C. How.,) tại Quảng Nam và Quảng Ninh bằng chỉ thị phân tử ISSR
Nghiên cứu được thực hiện nhằm đánh giá đa dạng di truyền của các cá thể ba kích trong các quần thể thu thập từ rừng trồng tự nhiên tại Tây Giang, tỉnh Quảng Nam và Ba Chẽ, tỉnh Quảng Ninh. Từ 11 mồi, chọn lọc được 6 mồi ISSR cho đa hình với số alen hiệu quả trung bình là 8,7. Kết quả đánh giá bằng chỉ thị ISSR có tổng số 52 alen từ 11 chỉ thị ISSR được phát hiện trên tổng số 45 cá thể thuộc ba quần thể nghiên cứu. Cây phân loại được xây dựng bằng phần mềm NTCYS 2.1 dựa trên hệ số tương đồng Jaccard, thuật toán UPGMA cho thấy 45 mẫu ba kích nghiên cứu được chia thành 2 nhóm lớn với hệ số tương đồng di truyền dao động trong khoảng 0,61 đến 1,0. Hệ số sai khác di truyền quần thể Nei Gst=0,3278 và hệ số trao đổi gen quần thể (gene flow) Nm= 1,0253. Điều này có thể khẳng định trong quần thể ba kích tự nhiên thuộc Quảng Nam và Quảng Ninh có sự đa dạng ở mức độ phân tử. Kết quả này sẽ hữu ích cho bảo tồn, chọn tạo ba kích tại tỉnh Quảng Nam và Quảng Ninh

Trang 1

Trang 2

Trang 3
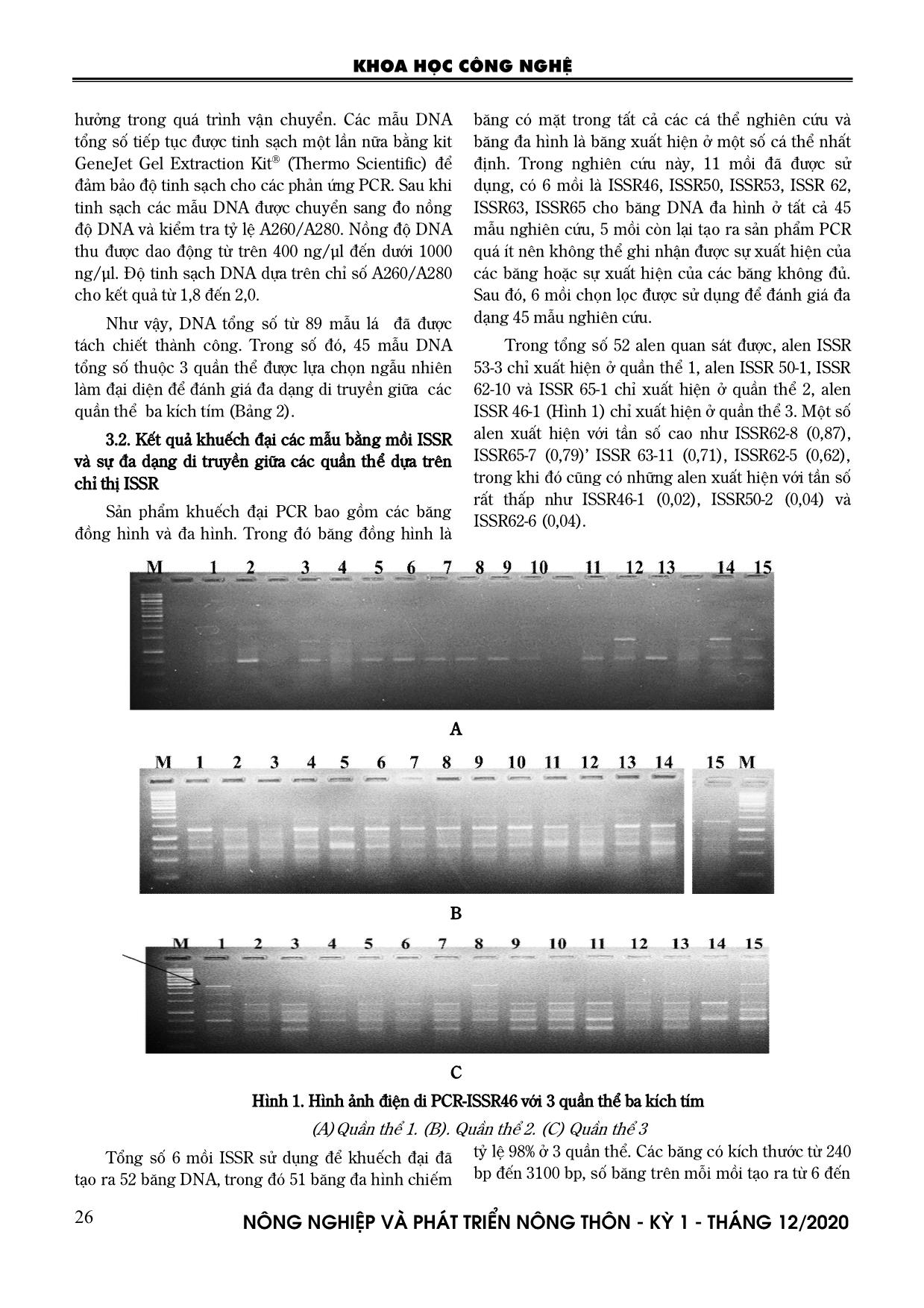
Trang 4
Trang 5

Trang 6

Trang 7

Trang 8
Tóm tắt nội dung tài liệu: Đánh giá đa dạng di truyền trong các quần thể ba kích tím (Morinda officinalis F. C. How.,) tại Quảng Nam và Quảng Ninh bằng chỉ thị phân tử ISSR

ược đánh số từ 1 đến 15, các mẫu thu tại xã A. Tiêng - huyện Tây Giang (quần thể 2) được đánh số từ 16 đến 30. Các mẫu thu tại xã Thanh Lâm, huyện Ba Chẽ, Quảng Ninh (quần thể 3) được sử dụng để so sánh, được đánh số từ 31 đến 45 (Bảng 2). Bảng 2. Danh sách các mẫu ba kích tím được sử dụng trong nghiên cứu STT Tên mẫu Địa điểm STT Tên mẫu Địa điểm STT Tên mẫu Địa điểm 1 1 16 16 31 31 2 2 17 17 32 32 3 3 18 18 33 33 4 4 19 19 34 34 5 5 20 20 35 35 6 6 21 21 36 36 7 7 22 22 37 37 8 8 23 23 38 38 9 9 24 24 39 39 10 10 25 25 40 40 11 11 26 26 41 41 12 12 27 27 42 42 13 13 28 28 43 43 14 14 29 29 44 44 15 15 Quần thể 1 Thôn A.Rớh – xã Lăng - huyện Tây Giang - Quảng Nam (132oĐN 15o51’8”B 107o28’3”Đ) 30 30 Quần thể 2 Xã A. Tiêng - huyện Tây Giang - Quảng Nam (358oB 15o52’49”B 107o31’24”Đ) 45 45 Quần thể 3 Xã Thanh Lâm - huyện Ba Chẽ, Quảng Ninh Kết quả điện di kiểm tra DNA tổng số trên gel agarose 1% cho thấy các mẫu DNA tổng số thu được có độ sáng đồng đều nhưng phần lớn xuất hiện nhiều đứt gãy (dải màu trắng chụp dưới đèn UV) có thể do các mẫu thu được là các lá ngoài tự nhiên và có chứa nhiều hợp chất thứ cấp hay chất lượng mẫu bị ảnh KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 1 - TH¸NG 12/2020 26 hưởng trong quá trình vận chuyển. Các mẫu DNA tổng số tiếp tục được tinh sạch một lần nữa bằng kit GeneJet Gel Extraction Kit® (Thermo Scientific) để đảm bảo độ tinh sạch cho các phản ứng PCR. Sau khi tinh sạch các mẫu DNA được chuyển sang đo nồng độ DNA và kiểm tra tỷ lệ A260/A280. Nồng độ DNA thu được dao động từ trên 400 ng/µl đến dưới 1000 ng/µl. Độ tinh sạch DNA dựa trên chỉ số A260/A280 cho kết quả từ 1,8 đến 2,0. Như vậy, DNA tổng số từ 89 mẫu lá đã được tách chiết thành công. Trong số đó, 45 mẫu DNA tổng số thuộc 3 quần thể được lựa chọn ngẫu nhiên làm đại diện để đánh giá đa dạng di truyền giữa các quần thể ba kích tím (Bảng 2). 3.2. Kết quả khuếch đại các mẫu bằng mồi ISSR và sự đa dạng di truyền giữa các quần thể dựa trên chỉ thị ISSR Sản phẩm khuếch đại PCR bao gồm các băng đồng hình và đa hình. Trong đó băng đồng hình là băng có mặt trong tất cả các cá thể nghiên cứu và băng đa hình là băng xuất hiện ở một số cá thể nhất định. Trong nghiên cứu này, 11 mồi đã được sử dụng, có 6 mồi là ISSR46, ISSR50, ISSR53, ISSR 62, ISSR63, ISSR65 cho băng DNA đa hình ở tất cả 45 mẫu nghiên cứu, 5 mồi còn lại tạo ra sản phẩm PCR quá ít nên không thể ghi nhận được sự xuất hiện của các băng hoặc sự xuất hiện của các băng không đủ. Sau đó, 6 mồi chọn lọc được sử dụng để đánh giá đa dạng 45 mẫu nghiên cứu. Trong tổng số 52 alen quan sát được, alen ISSR 53-3 chỉ xuất hiện ở quần thể 1, alen ISSR 50-1, ISSR 62-10 và ISSR 65-1 chỉ xuất hiện ở quần thể 2, alen ISSR 46-1 (Hình 1) chỉ xuất hiện ở quần thể 3. Một số alen xuất hiện với tần số cao như ISSR62-8 (0,87), ISSR65-7 (0,79)’ ISSR 63-11 (0,71), ISSR62-5 (0,62), trong khi đó cũng có những alen xuất hiện với tần số rất thấp như ISSR46-1 (0,02), ISSR50-2 (0,04) và ISSR62-6 (0,04). A B C Hình 1. Hình ảnh điện di PCR-ISSR46 với 3 quần thể ba kích tím (A) Quần thể 1. (B). Quần thể 2. (C) Quần thể 3 Tổng số 6 mồi ISSR sử dụng để khuếch đại đã tạo ra 52 băng DNA, trong đó 51 băng đa hình chiếm tỷ lệ 98% ở 3 quần thể. Các băng có kích thước từ 240 bp đến 3100 bp, số băng trên mỗi mồi tạo ra từ 6 đến KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 1 - TH¸NG 12/2020 27 11 băng với trung bình đạt 8,7/mồi. Mồi chỉ thị ISSR62 và ISSR63 cho nhiều băng đa hình nhất với 11 băng, mồi ISSR 46 và ISSR 50 cho số băng đa hình thấp nhất với 6 băng. Mồi ISSR 65 cho 8 băng, mồi ISSR 53 cho 10 băng. Tỷ lệ đa hình với các mồi ISSR trong nghiên cứu của chúng tôi cao hơn so với nghiên cứu của Hoàng Đăng Hiếu và cs (2016). Bảng 3. Biến động di truyền của ba quần thể ba kích dựa trên dữ liệu chỉ thị ISSR STT Tên mồi Kích thước băng (bp) Tổng số băng Số băng đa hình Tỷ lệ băng đa hình (%) 1 ISSR46 500-2700 6 5 83,3 2 ISSR50 500-1500 6 6 100 3 ISSR53 240-1600 10 10 100 4 ISSR62 950-3100 11 11 100 5 ISSR63 300-3000 11 11 100 6 ISSR65 560-3100 8 8 100 Tổng số băng 52 51 Trung bình 8,7 8,5 97,2 Bảng 4. Các chỉ số đa dạng trong 3 quần thể ba kích Tên quần thể NPB PPB (%) Na SD Ne SD h SD I SD QT1 37 71,15 1,9808 0,1387 1,5049 0,3624 0,2957 0,1708 0,4497 0,2181 QT2 22 42,31 1,4213 0,4989 1,2765 0,3664 0,1604 0,2017 0,2372 0,2913 QT3 36 69,23 1,6923 0,4660 1,3647 0,3589 0,2191 0,1897 0,3342 0,2683 Ghi chú: NPB: số phân đoạn đa hình. PPB(%): Phần trăm các băng DNA đa hình; Na: Số alen quan sát được; Ne: số alen có ý nghĩa; h: hệ số đa dạng di truyền Nei giữa các mẫu; I: hệ số Shannon; SD: độ lệch chuẩn. Số alen quan sát được (Na) của quần thể QT1 cao nhất đạt 1,9808 trong khi quần thể 3 có số alen quan sát được là 1,6923 và thấp nhất là quần thể 2, đạt 1,4213. Ngoài ra, số alen có ý nghĩa (Ne) biến động từ 1,2765 (quần thể 2) đến 1,5049 (quần thể 1) và trung bình là 1,3647 (quần thể 3). Trong nghiên cứu này, giá trị Na và Ne của quần thể 1 quan sát luôn lớn hơn và quần thể 2 có giá trị nhỏ nhất. Điều này chứng tỏ quần thể 1 có sự đa dạng lớn hơn quần thể 3 và quần thể 2 có mức độ đa dạng kém nhất. Chỉ số Na và Ne trong phân tích ở nghiên cứu này có giá trị lớn hơn so với nghiên cứu của Hiếu và cs (2016). Hệ số đa dạng di truyền Nei (h) trong ba quần thể trong khoảng từ 0,2957 đến 0,1604. Hệ số Shannon (I) trong khoảng từ 0,4497 đến 0,2372. Chỉ số sai khác di truyền Gst giữa các quần thể được tính toán sử dụng phần mềm POPGENE cho kết quả chỉ số Gst đạt 0,3278, chỉ ra mức độ sai khác thấp giữa các quần thể ba kích. Chỉ số Gst < 0,5 là kết quả của hầu hết các nghiên cứu ở các loài thực vật trong phân tích ISSR (Huang và cs, 2007; Yu và cs, 2011; Zhang và cs, 2010). Dựa trên giá trị Gst, giá trị Nm được tính toán và đạt 1,0253 cho thấy tần số trao đổi gen thấp giữa các quần thể (Bảng 5). Chỉ số sai khác di truyền giữa ba quần thể GST = 0,3278 chứng tỏ có 32,78% sự sai khác di truyền giữa các mẫu nghiên cứu. Mức độ trao đổi gen được thể hiện qua hệ số trao đổi gen (Nm) giữa ba quần thể thấp 1,0253. Hệ số trao đổi gen thấp giữa các quần thể là một đặc điểm chung ở các quần thể quý, hiếm có số lượng nhỏ. Ngược lại, với chọn tạo mẫu, sự biến động di truyền, sự đột biến, sự chọn lọc và cách ly di truyền sẽ tác động đến sự đa dạng nguồn gen. Bảng 5. Các chỉ số đa dạng giữa 3 các quần thể nghiên cứu Các thông số Mean ± SD Số lượng mẫu 45 HT 0,2957±0,0292 HS 0,1988±0,0141 GST 0,3278 Nm 1,0253 Ghi chú: HT: Chỉ số đa dạng nguồn gen của tổng các quần thể; HS: Chỉ số đa dạng gen trung bình trong quần thể; GST: Chỉ số sai khác di truyền Nei giữa các quần thể; Nm: Chỉ số trao đổi gen giữa các quần thể. KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 1 - TH¸NG 12/2020 28 Nhóm tác giả cũng tính toán hệ số tương đồng di tryền (Bảng 6) và xây dựng sơ đồ cây quan hệ di truyền (Hình 2) thể hiện mối quan hệ di truyền giữa các mẫu nghiên cứu. Bảng 6. Hệ số tương đồng di truyền giữa 45 cây ba kích nghiên cứu Hình 2. Cây quan hệ di truyền của các cá thể trong ba quần thể ba kích tím tại huyện Tây Giang, tỉnh Quảng Nam và huyện ba Chẽ, tỉnh Quảng Ninh KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 1 - TH¸NG 12/2020 29 Hệ số tương đồng di truyền phản ánh quan hệ di truyền của các cá thể ba kích khác nhau. Hai cá thể ba kích càng gần nhau về mặt di truyền thì hệ số tương đồng di truyền giữa chúng càng lớn và ngược lại. Kết quả phân tích tương đồng di truyền được thể hiện trên bảng 6. Hệ số tương đồng nội quần thể nằm trong khoảng 0,46 đến 1,00. Kết hợp với quan sát thực địa và nghiên cứu của Hoàng Đăng hiếu và cs (2016) có thể thấy sự suy thoái diện tích rừng ba kích ngoài tự nhiên và trồng mới tập trung hoặc phân tán là nguyên nhân dẫn đến sự giảm tính đa dạng di truyền quần thể. So sánh sự tương đồng di truyền giữa cả ba quần thể khảo sát cũng cho kết quả tương tự. Dựa vào cây phân loại được xây dựng dựa trên các dữ liệu về khoảng cách di truyền có thể thấy rõ ràng sự phân chia thành hai nhánh lớn riêng biệt: Nhánh 1 bao gồm các cá thể thuộc quần thể 1 và 2; Nhánh 2 bao gồm các cá thể thuộc quần thể 3. Kết quả này cũng phù hợp với phân bố tự nhiên của các quần thể trong nghiên cứu: quần thể 1 và 2 là 2 quần thể có phân bố tại huyện Tây Giang, tỉnh Quảng Nam, còn quần thể 3 là thuộc huyện Ba Chẽ, tỉnh Quảng Ninh và hai tỉnh này có vị trí địa lý phân bố rất xa nhau. 4. KẾT LUẬN VÀ ĐỀ NGHỊ Như vậy, trong tổng số 11 mồi ISSR được sử dụng trong nghiên cứu có 6 mồi tạo ra các băng đa hình, với số alen trung bình là 8,7/mồi và 45 mẫu nguồn gen phân thành 2 nhóm lớn với hệ số tương đồng di truyền dao động trong khoảng 0,46 đến 1,0. Trong đó nhóm lớn 1 bao gồm các mẫu thuộc quần thể 1 (thôn A.Rớh - xã Lăng - huyện Tây Giang) và phần lớn các mẫu thuộc quần thể 2 (xã A. Tiêng - huyện Tây Giang, tỉnh Quảng Nam) và nhóm lớn 2 bao gồm các mẫu thuộc huyện Ba Chẽ, tỉnh Quảng Ninh và hai mẫu 29, 30 của quần thể 2. Dữ liệu được phân tích trong nghiên cứu này sẽ cung cấp thông tin hữu ích trong việc khảo sát, phân tích hiện trạng và đa dạng của quần thể ba kích tím tại Quảng Nam, từ đó góp phần chọn tạo và bảo tồn cây ba kích tím tại huyện Tây Giang, tỉnh Quảng Nam. LỜI CẢM ƠN Công trình này được thực hiện nhờ kinh phí Dự án:“Phát triển nguồn dược liệu ba kích tím Morinda officinalis tại Hiệp Đức, Quảng Nam phục vụ chế biến thuốc và thực phẩm chức năng” mã số VAST.SXTN-06/17-19. TÀI LIỆU THAM KHẢO 1. Ding P, Liu J, Yang TC (2008). Genetic diversity of Morinda officinalis by RAPD. Chin Trad Herb Drug 39:1869–1872. 2. Đinh Thị Phòng, Nguyễn Thị Liễu, Vũ Thị Thu Hiền, Trần Thị Liễu, Trần Thị Việt Thanh, Nguyễn Quốc Bình, Vũ Đình Duy, Nguyễn Tiến Hiệp, Phạm Hữu Nhân (2015). Đánh giá đa dạng di truyền quần thể tự nhiên loài Kim giao núi đất (Nageia wallichiana (C. Presl) Kuntze) ở Tây Nguyên bằng chỉ thị ISSR. Tạp chí Công nghệ Sinh học 13(1): 131–141. 3. Doyle J. J, Doyle J. J (1989). A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical Bulletin 19: 11-15. 4. Hoàng Đăng Hiếu, Chu Thị Thu Hà, Phạm Bích Ngọc, Lâm Đại Nhân, Nguyễn Thị Thúy Hường, Chu Hoàng Hà (2016). Sử dụng chỉ thị ISSR trong việc đánh giá đa dạng di truyền ở quần thể ba kích tại Quảng Ninh. Tạp chí Sinh học 38(1): 89-95. 5. Li B, Lu C, Zhou ZY, Xiang ZH (2000). Construction of silkworm RAPD molecular linkage map. Yi Chuan Xue Bao 27(2):127-132. 6. Liu Y-J, Huang Y, Rong J-D, Zhang Y, Xue Y- M, Zheng Y-S (2011). Genetic diversity analysis of Morinda officinalis by ISSR markers. Journal of Fujian College of Forestry 31(3):203–206. 7. Huang N-Z, Fu C-M, Zhao Z-G, Tang F-L, Li F (2007). Tissue culture and rapid proliferation of Morinda officinalis How. Botany, Guangxi Zhuangzu Autonomous Region and the Chinese Academy of Sciences, Guilin 541006, China 27(1):127-131. 8. Nguyễn Đức Thành (2014). Các kỹ thuật chỉ thị DNA trong nghiên cứu và chọn lọc thực vật. Tạp chí Sinh học 36(3): 265-294. 9. Pillai P. P, Sajan S. J, Menon M. K, Jayakumar P. S K, Subramoniam A (2012). ISSR analysis reveals high intraspecific variation in Rauvolfia serpentina L. - A high value medicinal plant. Biochemical Systematics and Ecology, 40: 192- 197. 10. Trần Thị Liễu, Vũ Thị Thu Hiền, Nguyễn Tiến Hiệp, Đinh Thị Phòng (2015). Tính đa dạng nguồn gen di truyền loài Thông lá dẹt (Pinus krempfii Lecomte) - Loài đặc hữu hẹp ở Tây Nguyên, Việt Nam bằng chỉ thị ISSR. Tạp chí Khoa học và Công nghệ 53 (2): 169–179. KHOA HỌC CÔNG NGHỆ N«ng nghiÖp vµ ph¸t triÓn n«ng th«n - KỲ 1 - TH¸NG 12/2020 30 11. Wang D. X, Wang J, Ding S. X, Su Y. Q. (2007). Comparative studies on some genetic characteristics among four large yellow croaker (Pseudosciaena crocea) populations. Acta Oceanologica Sinica 26(4): 148-155. 12. Wei LJ, Lu P and Su WP. (2006). Tissue culture and rapid propagation of Morinda officinalis How. Plant Physilogy Comunication 42 (3): 475. 13. Wu ZQ, Chen DL, Lin FH. (2015). Effect of bajijiasu isolated from Morinda officinalis F. C. how on sexual function in male mice and its antioxidant protection of human sperm. J Ethnopharmacol 164:283–292. 14. Yeh FC, Yang R.C, Boyle T.BJ, Ye ZH, Mao JX (1997). POPGENE, the User Friendly Shareware for Population Genetic Analysis. Molecular Biology and Biotechnology Centre, University of Alberta, Canada. 15. Yu HH, Yang LZ, Sun B, Liu NR (2011). Genetic diversity and relationships of endangered plant Magnolia officinalis (Magnoliaceae) assessed with ISSR polymorphisms. Biochemical Systematics and Ecology 39(2): 71-78. 16. Zhang JH, Xin HL, Xu YM (2018). Morinda officinalis How. A comprehensive review of traditional uses, phytochemistry and pharmacology. J Ethnopharmacol 213:230–255. 17. Zhang QD, Gao ML, Yang P. Y (2010). Genetic diversity and structure of a traditional Chinese medicinal plant species, Fritillaria cirrhosa (Liliaceae) in Southwest China and implications for its conservation. Biochemical Systematics and Ecology 38(3): 236-242. GENETIC DIVERSITY IN Morinda officinalis F. C. How POPULATIONS IN QUANG NAM AND QUANG NINH PROVINCES USING ISSR MARKERS Tran Thi Huong Giang , Nguyen Thi Thuy Huong, Nguyen Thi Thu Hien, Tran Ho Quang, Nguyen Duc Thuan, Pham Bich Ngoc Summary The purpose of this study was to analyze genetic diversity of Morinda officinalis populations collected from Tay Giang, Quang Nam province and Ba Che - Quang Ninh province. Of the 11 ISSR primers screened, 6 primers produced highly reproducible bands with an average of 8.7 alleles per locus. Evaluation of genetic diversity of 45 individuals with ISSR primers, a total of 52 alleles were identified using 6 selected primers. The phylogenetic tree constructed by NTSYS PC 2.1 based on Jaccard's genetic coefficient using the algorithm UPGMA showed two major groups ranging from 0.61 to 1.0. The result revealed the value of Nei’s genetic differentiation index (GST) and the estimated gene flow (Nm), which were about 0.3278 and 1.0253, respectively. These results demonstrated the diversity state of M. officinalis populations distributed in Quang Nam and Quang Ninh. These achievements was useful for conservation and breeding, of Morinda officinalis How., Keywords: ISSR marker, genetic diversity, Morinda officinalis How., Quang Nam. Người phản biện: TS. Nguyễn Văn Khiêm Ngày nhận bài: 10/7/2020 Ngày thông qua phản biện: 10/8/2020 Ngày duyệt đăng: 17/8/2020
File đính kèm:
 danh_gia_da_dang_di_truyen_trong_cac_quan_the_ba_kich_tim_mo.pdf
danh_gia_da_dang_di_truyen_trong_cac_quan_the_ba_kich_tim_mo.pdf

